
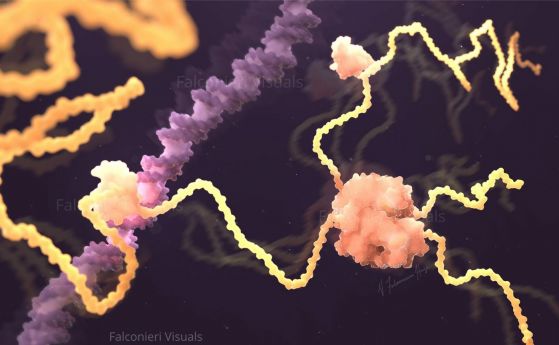
С помощта на изкуствен интелект изследователи са успели да разкрият 70 500 вируса, неизвестни досега на науката, много от тях са странни и по нищо не приличат на известни видове.
РНК вирусите са идентифицирани с помощта на метагеномика, при която учените правят проби от всички геноми, присъстващи в околната среда, без да се налага да култивират отделни вируси. Методът показва потенциала на AI да изследва "тъмната материя" на вселената на РНК вируса.
Вирусите са микроорганизми, които заразяват животни, растения и дори бактерии, но само малка част от тях са идентифицирани и описани. По същество има "бездънна яма" от вируси за откриване, коментира Артьом Бабаян (Artem Babaian), компютърен вирусолог от Университета в Торонто, Канада. Някои от тези вируси биха могли да причинят заболявания при хората, което означава, че определянето на характеристиките им може да помогне за обяснението на загадъчни заболявания, посочва Бабаян.
В предишни проучвания е използвано машинно обучение за откриване на нови вируси в данните от секвенирането. Последното проучване, публикувано в Cell миналата седмица, прави още една стъпка напред в тази работа и я използва за анализиране на предсказани протеинови структури.
Моделът на изкуствения интелект включва инструмент за прогнозиране на протеини, наречен ESMFold, който е разработен от изследователи от Meta (бивш Facebook, със седалище в Менло Парк, Калифорния). Подобна система за AI, AlphaFold, е разработена от изследователи в Google DeepMind в Лондон, които тази седмица спечелиха Нобелова награда за химия.
Пропуснати вируси
През 2022 г. Бабаян и колегите му претърсват 5,7 милиона геномни проби, архивирани в публично достъпни бази данни, и идентифицират почти 132 000 нови РНК вируси. И други групи са ръководили подобни изследвания.
Но РНК вирусите еволюират бързо, така че съществуващите методи за идентифициране на РНК вируси в данни за геномни последователности вероятно пропускат много от тях. Обичайният метод е да се търси участък от генома, който кодира ключов протеин, използван при репликацията на РНК, наречен РНК-зависима РНК-полимераза (RdRp - RNA-dependent RNA polymerase). Но ако последователността, която кодира този протеин в даден вирус, се различава значително от всички известни последователности, изследователите няма да го разпознаят.
Шъ Ман (Shi Mang), еволюционен биолог от университета "Сун Ят-сен" в Шънджън, Китай, и съавтор на изследването в Cell, и колегите му търсят неразпознати досега вируси в публично достъпни геномни проби.
Те разработват модел, наречен LucaProt, използвайки архитектурата "трансформатор", която е в основата на ChatGPT, и му предоставят данни за секвениране и предсказване на протеини от ESMFold. След това обучават модела си да разпознава вирусни RdRps и го използват, за да намерят в голямата партида данни от геномни секвенции, които кодират тези ензими - доказателство, че тези последователности принадлежат на вирус. Използвайки този метод, те идентифицирали около 160 000 РНК-вируса, включително някои от тях, които били изключително дълги и се срещали в екстремни среди като горещи извори, солени езера и въздух. Малко по-малко от половината от тях не са били описани досега. Те откриват "малки джобове на биоразнообразие от РНК вируси, които са наистина далеч в еволюционното пространство", посочва Бабаян.
"Това е наистина обещаващ подход за разширяване на виросферата", коментира Джаки Махар (Jackie Mahar), еволюционен вирусолог в Австралийския център за готовност за болести CSIRO в Гелонг.
Описанието на вирусите ще помогне на изследователите да разберат произхода на микробите и как те са еволюирали в различните гостоприемници, добавя Махар.
А разширяването на кръга от познати вируси улеснява намирането на още подобни вируси, отбелязва Бабаян.
"Изведнъж можете да видите неща, които просто не сте виждали преди."
Екипът не е успял да определи гостоприемниците на идентифицираните от него вируси, което трябва да се проучи допълнително, уточнява Махар. Изследователите са особено заинтересовани да разберат дали някой от новите вируси заразява археи - цял клон от дървото на живота, за който няма ясно доказано, че се заразява от РНК вируси.
Сега Шъ разработва модел за прогнозиране на гостоприемниците на тези новоидентифицирани РНК вируси. Той се надява, че това ще помогне на изследователите да разберат ролята, която вирусите имат в техните екологични ниши.
Справка:
-
Hou, X. et al. Cell https://doi.org/10.1016/j.cell.2024.09.027 (2024).
-
Edgar, R. C. et al. Nature 602, 142–147 (2022).
-
Zayed, A. A. et al. Science 376,156–162 (2022).
Източник: AI scans RNA ‘dark matter’ and uncovers 70,000 new viruses, Smriti Mallapaty, Nature, doi: https://doi.org/10.1038/d41586-024-03320-6
Още по темата

Физика
Нобелова награда за химия 2024 за разкриването на тайните на протеините чрез изчисления и изкуствен интелект

Човекът
Списание Science обяви 10-те най-големи научни пробива за 2022 г.
Животът
Наистина ли AlphaFold е решил проблема на биологията с нагъването на протеините?

















Коментари
Моля, регистрирайте се от TУК!
Ако вече имате регистрация, натиснете ТУК!
Няма коментари към тази новина !
Последни коментари
4I4ATA
Нов, скрит досега вход в пирамидата на Микерин е открит по аномалии при сканиране
YKoshev
Престижна награда от БАН спечели главният редактор на НаукаOFFNews
Johnny B Goode
Престижна награда от БАН спечели главният редактор на НаукаOFFNews
Gunteer
Престижна награда от БАН спечели главният редактор на НаукаOFFNews