
–ù–Α –Κ―Ä–Α―è –Ϋ–Α –≤―¹―è–Κ–Α –≥–Ψ–¥–Η–Ϋ–Α ―¹–Ω–Η―¹–Α–Ϋ–Η–Β Science –Ψ–Ω―Ä–Β–¥–Β–Μ―è –Κ–Ψ–Η ―¹–Α –±–Η–Μ–Η –Ϋ–Α–Ι-–Ζ–Ϋ–Α―΅–Η–Φ–Η―²–Β –Ψ―²–Κ―Ä–Η―²–Η―è. –ü―Ä–Α–≤―è―² –Η –¥–Ψ–Ω–Η―²–≤–Α–Ϋ–Β –¥–Ψ ―΅–Η―²–Α―²–Β–Μ–Η―²–Β ―¹–Η. –€–Ϋ–Β–Ϋ–Η―è―²–Α –Ϋ–Α ―Ä–Β–¥–Α–Κ―Ü–Η―è―²–Α –Η –Ϋ–Α ―΅–Η―²–Α―²–Β–Μ–Η―²–Β ―¹―ä–≤–Ω–Α–¥–Α―² –Ζ–Α –Ω―ä―Ä–≤–Η –Ω―ä―² –Ψ―² 2018 –≥.
–û―²–Κ―Ä–Ψ―è–≤–Α―² ―¹–Β ―²―Ä–Η–Φ–Α ―³–Η–Ϋ–Α–Μ–Η―¹―²–Η:
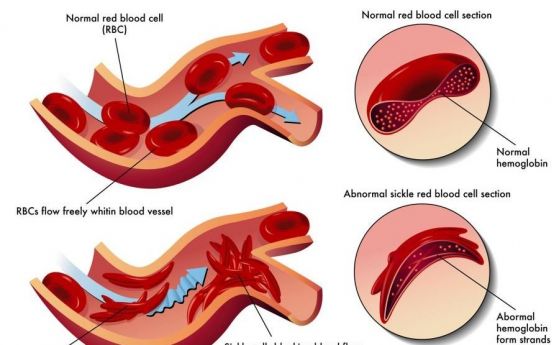
1. –‰–Ζ–Ω–Ψ–Μ–Ζ–≤–Α–Ϋ–Β―²–Ψ –Ϋ–Α –î–ù–ö –≤ –¥―Ä–Β–≤–Ϋ–Α –Κ–Α–Μ (–≤–Β―΅–Β –Ϋ―è–Φ–Α –Ϋ―É–Ε–¥–Α –Η–Ζ –Ω–Β―â–Β―Ä–Η―²–Β –¥–Α ―¹–Β ―²―ä―Ä―¹–Η –Ζ―ä–±―΅–Β –Η–Μ–Η –Κ–Ψ―¹―²–Η―΅–Κ–Α –Ζ–Α –î–ù–ö –Α–Ϋ–Α–Μ–Η–Ζ, –¥–Ψ―¹―²–Α―²―ä―΅–Ϋ–Ψ –Β –¥–Α ―¹–Β –≥―Ä–Β–±–Ϋ–Β –Φ–Α–Μ–Κ–Ψ –Ω–Β―â–Β―Ä–Ϋ–Α –Κ–Α–Μ, –Η –Ψ―² –Α–Ϋ–Α–Μ–Η–Ζ–Α –Ϋ–Α –î–ù–ö –≤ –Ϋ–Β―è ―â–Β ―¹–Β –Ϋ–Α―É―΅–Α―² –Ϋ–Β―â–Α –Ζ–Α –Ψ―²–¥–Α–≤–Ϋ–Α―à–Ϋ–Η –Ψ–±–Η―²–Α―²–Β–Μ–Η –Ϋ–Α –¥―Ä–Β–≤–Ϋ–Η –Ω–Β―â–Β―Ä–Η, –Ζ–Α –¥–Η–Β―²–Α―²–Α –Η–Φ –Η –Ω―Ä–Ψ―΅–Β–Β); (–ü–Ψ–≤–Β―΅–Β –Η–Ϋ―³–Ψ―Ä–Φ–Α―Ü–Η―è "–‰–Ζ–≤–Μ–Β―΅–Β–Ϋ–Α –Β –î–ù–ö –Ϋ–Α –¥―Ä–Β–≤–Ϋ–Η ―Ö–Ψ―Ä–Α –Ζ–Α –Ω―ä―Ä–≤–Η –Ω―ä―² –Ψ―² –Ω–Β―â–Β―Ä–Ϋ–Η –Ω–Ψ―΅–≤–Η".
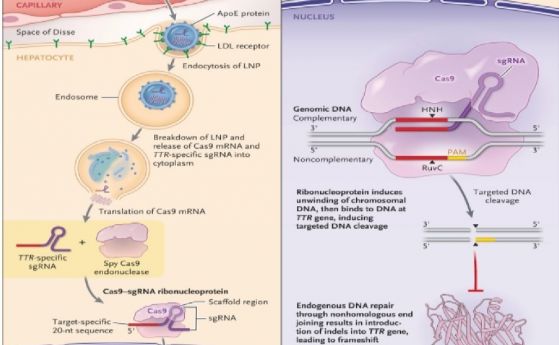
2. –Θ―¹–Ω–Β―à–Ϋ–Ψ―²–Ψ –Ω―Ä–Η–Μ–Α–≥–Α–Ϋ–Β –Ϋ–Α –≥–Β–Ϋ–Ϋ–Ψ―²–Ψ ―Ä–Β–¥–Α–Κ―²–Η―Ä–Α–Ϋ–Β ―΅―Ä–Β–Ζ CRISPR –Ϋ–Α –Φ―è―¹―²–Ψ –≤ ―΅–Ψ–≤–Β―à–Κ–Ψ―²–Ψ ―²―è–Μ–Ψ –Ζ–Α –Μ–Β–Κ―É–≤–Α–Ϋ–Β –Ϋ–Α –Ϋ–Α―¹–Μ–Β–¥―¹―²–≤–Β–Ϋ–Η –±–Ψ–Μ–Β―¹―²–Η (―¹―ä―Ä–Ω–Ψ–≤–Η–¥–Ϋ–Α –Α–Ϋ–Β–Φ–Η―è –Η –±–Β―²–Α-―²–Α–Μ–Α―¹–Β–Φ–Η―è ("–™–Β–Ϋ–Ϋ–Η ―²–Β―Ä–Α–Ω–Η–Η –Ζ–Α –Μ–Β–Κ―É–≤–Α–Ϋ–Β –Ϋ–Α ―¹―ä―Ä–Ω–Ψ–≤–Η–¥–Ϋ–Ψ-–Κ–Μ–Β―²―ä―΅–Ϋ–Α –Α–Ϋ–Β–Φ–Η―è –Η –±–Β―²–Α-―²–Α–Μ–Α―¹–Β–Φ–Η―è") ―²―Ä–Α–Ϋ―¹―²–Η―Ä–Β―²–Η–Ϋ (TTR) –Α–Φ–Η–Μ–Ψ–Η–¥–Ψ–Ζ–Α (–™–Β–Ϋ–Ϋ–Α ―²–Β―Ä–Α–Ω–Η―è ―¹ CRISPR ―΅―Ä–Β–Ζ ―²―Ä–Α–Ϋ―¹―³―É–Ζ–Η―è –Ϋ–Α –†–ù–ö –≤ –Κ―Ä―ä–≤―²–Α); –Ζ―Ä–Η―²–Β–Μ–Β–Ϋ –¥–Β―³–Β–Κ―² –¦–Β–±–Β―Ä –Κ–Ψ–Ϋ–≥–Β–Ϋ–Η―²–Α–Μ–Ϋ–Α –Α–Φ–Α―É―Ä–Ψ–Ζ–Α 10);
3. –Θ–¥–Η–≤–Η―²–Β–Μ–Ϋ–Ψ ―²–Ψ―΅–Ϋ–Ψ –Ω―Ä–Β–¥―¹–Κ–Α–Ζ–≤–Α–Ϋ–Β –Ϋ–Α –±–Β–Μ―²―ä―΅–Ϋ–Η ―¹―²―Ä―É–Κ―²―É―Ä–Η ―¹ –Ω–Ψ–Φ–Ψ―â―²–Α –Ϋ–Α –Φ–Β―²–Ψ–¥–Η–Κ–Η –Ϋ–Α –Η–Ζ–Κ―É―¹―²–≤–Β–Ϋ –Η–Ϋ―²–Β–Μ–Β–Κ―².
–ü―ä―Ä–≤–Ψ –Φ―è―¹―²–Ψ ―¹–Ω–Β―΅–Β–Μ–≤–Α –Η–Ζ–Κ―É―¹―²–≤–Β–Ϋ–Η―è―² –Η–Ϋ―²–Β–Μ–Β–Κ―² (–‰–‰) βÄ™ –Ζ–Α –Ϋ–Α–Ι-–≤–Ω–Β―΅–Α―²–Μ―è–≤–Α―â–Ψ―²–Ψ –Ζ–Α–≤–Ψ–Β–≤–Α–Ϋ–Η–Β: –ù–Β ―¹–Α–Φ–Ψ –Ζ–Α –Ω―Ä–Β–¥―¹–Κ–Α–Ζ–≤–Α–Ϋ–Β –Ϋ–Α –±–Β–Μ―²―ä―΅–Ϋ–Η ―¹―²―Ä―É–Κ―²―É―Ä–Η, –Ϋ–Ψ –Η –Ζ–Α –Ϋ–Α–Φ–Η―Ä–Α–Ϋ–Β –Ϋ–Α –Ϋ–Α―΅–Η–Ϋ–Α, –Ω–Ψ –Κ–Ψ–Ι―²–Ψ ―²–Β ―¹–Η –≤–Ζ–Α–Η–Φ–Ψ–¥–Β–Ι―¹―²–≤–Α―² –Η –Ψ–±―Ä–Α–Ζ―É–≤–Α―² –Κ–Ψ–Φ–Ω–Μ–Β–Κ―¹–Η. –ü–Ψ―¹–Μ–Β–¥–Ϋ–Α―²–Α βÄû―΅–Β―Ä–Β―à–Κ–ΑβÄ€ –≤―ä―Ä―Ö―É ―²–Ψ―Ä―²–Α―²–Α –Β ―¹―²–Α―²–Η―è –≤ ―¹–Ω. Nature –Ψ―² 16 –¥–Β–Κ–Β–Φ–≤―Ä–Η 2021, –≤ –Κ–Ψ―è―²–Ψ ―¹–Β –Ψ–Ω–Η―¹–≤–Α –Κ–Α–Κ ―¹ –‰–‰ –Φ–Ψ–Ε–Β –¥–Α ―¹–Β –Κ–Ψ–Ϋ―¹―²―Ä―É–Η―Ä–Α―² –Ϋ–Ψ–≤–Η –±–Β–Μ―²―ä―Ü–Η –Κ–Α―²–Ψ ―¹–Β –Η–Ζ–Ω–Ψ–Μ–Ζ―É–≤–Α –Φ–Β―²–Ψ–¥–Α –Ϋ–Α βÄû―Ö–Α–Μ―é―Ü–Η–Ϋ–Α―Ü–Η―èβÄ€, –Η–Ζ–Ω–Ψ–Μ–Ζ―É–≤–Α―â –Ω–Μ–Α―²―³–Ψ―Ä–Φ–Η –Ϋa Google DeepDream –Ζ–Α ―²―Ä–Β–Ϋ–Η―Ä–Α–Ϋ–Β –Ω―Ä–Η ―Ä–Α–Ζ–Ω–Ψ–Ζ–Ϋ–Α–≤–Α–Ϋ–Β –Ϋ–Α –Μ–Η―Ü–Α.

–‰–Ζ–Κ―É―¹―²–≤–Β–Ϋ–Η―è―² –‰–Ϋ―²–Β–Μ–Β–Κ―² –Ω―Ä–Β–¥―¹–Κ–Α–Ζ–≤–Α –Κ–Α–Κ –¥–≤–Α –±–Β–Μ―²―ä–Κ–Α –Ψ–±―Ä–Α–Ζ―É–≤–Α―² –Κ–Ψ–Φ–Ω–Μ–Β–Κ―¹, –Η–Ζ–≤―ä―Ä―à–≤–Α―â –Ω–Ψ–Ω―Ä–Α–≤–Κ–Η –Ϋ–Α –î–ù–ö –Ω―Ä–Η –¥―Ä–Ψ–Ε–¥–Η
CREDIT: I. R. HUMPHREYS ET AL., SCIENCE 374, 6573, EABM4805 (2021) DOI:10.1126/SCIENCE.ABM4805
–‰―¹―²–Ψ―Ä–Η―è―²–Α –Ϋ–Α –±–Β–Μ―²―ä―΅–Ϋ–Η―²–Β ―¹―²―Ä―É–Κ―²―É―Ä–Η –Ζ–Α–Ω–Ψ―΅–≤–Α ―¹ ―Ä–Α–Ζ―à–Η―³―Ä–Ψ–≤–Α–Ϋ–Β –Ϋ–Α ―Ä–Β–Ϋ―²–≥–Β–Ϋ–Ψ–≤–Α―²–Α –Κ―Ä–Η―¹―²–Α–Μ–Ψ–≥―Ä–Α–Φ–Α –Ϋ–Α –Φ–Η–Ψ–≥–Μ–Ψ–±–Η–Ϋ–Α, –Ζ–Α –Κ–Ψ–Β―²–Ψ –€–Α–Κ―¹ –ü–Β―Ä―É―Ü –Η –î–Ε–Ψ–Ϋ –ö–Β–Ϋ–¥―Ä―é –Ω–Ψ–Μ―É―΅–Α–≤–Α―² –ù–Ψ–±–Β–Μ–Ψ–≤–Α –Ϋ–Α–≥―Ä–Α–¥–Α. –ù–Β –¥―ä–Μ–≥–Ψ ―¹–Μ–Β–¥ ―²–Ψ–≤–Α –ö―Ä–Η―¹―²–Η―è–Ϋ –ê–Ϋ―³–Η–Ϋ―¹–Β–Ϋ ―É―¹―²–Α–Ϋ–Ψ–≤–Η, ―΅–Β –Φ–Ψ–Ε–Β –¥–Α ―Ä–Α–Ζ–≤–Α–Μ–Η, –Α ―¹–Μ–Β–¥ ―²–Ψ–≤–Α –¥–Α ―¹–Β –≤―ä–Ζ―¹―²–Α–Ϋ–Ψ–≤–Η, –Ω―Ä–Ψ―¹―²―Ä–Α–Ϋ―¹―²–≤–Β–Ϋ–Α―²–Α ―¹―²―Ä―É–Κ―²―É―Ä–Α –Η –±–Η–Ψ―Ö–Η–Φ–Η―΅–Ϋ–Α –Α–Κ―²–Η–≤–Ϋ–Ψ―¹―² –Ϋ–Α –Β–Ϋ–Ζ–Η–Φ–Α ―Ä–Η–±–Ψ–Ϋ―É–Κ–Μ–Β–Α–Ζ–Α. –û―² ―²–Ψ–≤–Α ―¹–Μ–Β–¥–≤–Α –≤–Α–Ε–Ϋ–Ψ―²–Ψ –Ζ–Α–Κ–Μ―é―΅–Β–Ϋ–Η–Β, ―΅–Β –Ζ–Α –Ω―Ä–Α–≤–Η–Μ–Ϋ–Ψ―²–Ψ –Ζ–Α–≥―ä–≤–Α–Ϋ–Β –Ϋ–Α –±–Β–Μ―²―ä–Κ–Α –≤ –Ω―Ä–Ψ―¹―²―Ä–Α–Ϋ―¹―²–≤–Ψ―²–Ψ –Β –Ϋ―É–Ε–Ϋ–Α ―¹–Α–Φ–Ψ –Ϋ–Β–≥–Ψ–≤–Α―²–Α –Ω―ä―Ä–≤–Η―΅–Ϋ–Α ―¹―²―Ä―É–Κ―²―É―Ä–Α - –Ω–Ψ―¹–Μ–Β–¥–Ψ–≤–Α―²–Β–Μ–Ϋ–Ψ―¹―²―²–Α –Ψ―² –Α–Φ–Η–Ϋ–Ψ–Κ–Η―¹–Β–Μ–Η–Ϋ–Η –Ω–Ψ –Ω–Ψ–Μ–Η–Ω–Β–Ω―²–Η–¥–Ϋ–Α―²–Α –≤–Β―Ä–Η–≥–Α. –Δ–Ψ–≤–Α ―Ä–Β–Ψ–Ω–Α–Κ–Ψ–≤–Α–Ϋ–Β ―¹―²–Α–≤–Α –±―ä―Ä–Ζ–Ψ - –Ζ–Α –Ϋ―è–Κ–Ψ–Μ–Κ–Ψ –Φ–Η–Κ―Ä–Ψ –¥–Ψ –Φ–Η–Μ–Η―¹–Β–Κ―É–Ϋ–¥–Η. –ö―Ä–Α–Ι–Ϋ–Α―²–Α ―¹―²–Α–±–Η–Μ–Ϋ–Α ―¹―²―Ä―É–Κ―²―É―Ä–Α –Β –≤ –Ψ–Ω―²–Η–Φ–Α–Μ–Β–Ϋ –Β–Ϋ–Β―Ä–≥–Β―²–Η―΅–Β–Ϋ –Φ–Η–Ϋ–Η–Φ―É–Φ.
–î–Ψ―¹–Β–≥–Α ―¹–Α –Ψ―²–Κ―Ä–Η―²–Η –Β–Κ―¹–Ω–Β―Ä–Η–Φ–Β–Ϋ―²–Α–Μ–Ϋ–Ψ ―¹―²―Ä―É–Κ―²―É―Ä–Η―²–Β –Ϋ–Α 185000 –±–Β–Μ―²―ä―Ü–Η, –Κ–Α―²–Ψ –Ζ–Α ―Ü–Β–Μ―²–Α ―²–Β ―¹–Β –Κ―Ä–Η―¹―²–Α–Μ–Η–Ζ–Η―Ä–Α―² –Η –Ψ–±–Μ―ä―΅–≤–Α―² ―¹ ―Ä–Β–Ϋ―²–≥–Β–Ϋ–Ψ–≤–Η –Μ―ä―΅–Η. –ö―Ä–Η―¹―²–Α–Μ–Η–Ζ–Η―Ä–Α–Ϋ–Β―²–Ψ –Β ―²―Ä―É–¥–Ψ–Β–Φ―ä–Κ –Ω―Ä–Ψ―Ü–Β―¹. –ù–Α –Ω–Ψ–Φ–Ψ―â –Η–¥–≤–Α –Φ–Β―²–Ψ–¥―ä―² –Ϋ–Α –Κ―Ä–Η–Ψ–Β–Μ–Β―²―Ä–Ψ–Ϋ–Ϋ–Α―²–Α –Φ–Η–Κ―Ä–Ψ―¹–Κ–Ψ–Ω–Η―è, –Ω―Ä–Η –Κ–Ψ–Ι―²–Ψ –Ϋ–Β –Β –Ϋ―É–Ε–Ϋ–Α –Κ―Ä–Η―¹―²–Α–Μ–Η–Ζ–Α―Ü–Η―è, –Ϋ–Ψ –Ζ–Α―²–Ψ–≤–Α –Ω―ä–Κ ―¹–Β –Η–Ζ–Ω–Ψ–Μ–Ζ–≤–Α –Φ–Ϋ–Ψ–≥–Ψ ―¹–Κ―ä–Ω –Η–Ϋ―¹―²―Ä―É–Φ–Β–Ϋ―²–Α―Ä–Η―É–Φ. –‰–Ζ–Ω―Ä–Α–≤–Β–Ϋ–Η –Ω―Ä–Β–¥ –Φ–Η–Μ–Η–Ψ–Ϋ–Η―²–Β –±–Β–Μ―²―ä―Ü–Η, –Κ–Ψ–Η―²–Ψ ―¹―ä―â–Β―¹―²–≤―É–≤–Α―² –≤ –Ε–Η–≤–Η―è ―¹–≤―è―², ―²―Ä―è–±–≤–Α –¥–Α ―¹–Β ―²―ä―Ä―¹–Η –¥―Ä―É–≥–Ψ ―Ä–Β―à–Β–Ϋ–Η–Β –Κ–Α―²–Ψ ―¹–Β –Ω―Ä–Β–¥―¹–Κ–Α–Ζ–≤–Α ―¹―²―Ä―É–Κ―²―É―Ä–Α―²–Α –Ϋ–Α –±–Β–Μ―²―ä–Κ–Α –Ω–Ψ –Ϋ–Β–≥–Ψ–≤–Α―²–Α –Ω―ä―Ä–≤–Η―΅–Ϋ–Α –Ω–Ψ―¹–Μ–Β–¥–Ψ–≤–Α―²–Β–Μ–Ϋ–Ψ―¹―² –Ψ―² –Α–Φ–Η–Ϋ–Ψ–Κ–Η―¹–Β–Μ–Η–Ϋ–Η.
–û―â–Β –Ω―Ä–Β–¥–Η –Ω–Ψ–Μ–Ψ–≤–Η–Ϋ –≤–Β–Κ –Ζ–Α–Ω–Ψ―΅–≤–Α―² –Ψ–Ω–Η―²–Η―²–Β –Ζ–Α –Ϋ–Α–Φ–Η―Ä–Α–Ϋ–Β –Ϋ–Α –Ω―Ä–Ψ―¹―²―Ä–Α–Ϋ―¹―²–≤–Β–Ϋ–Α―²–Α ―¹―²―Ä―É–Κ―²―É―Ä–Α –Ϋ–Α –±–Β–Μ―²―ä―Ü–Η ―΅―Ä–Β–Ζ –Φ–Ψ–¥–Β–Μ–Η―Ä–Α–Ϋ–Β ―¹ –Ω–Ψ–Φ–Ψ―â―²–Α –Ϋ–Α –Κ–Ψ–Φ–Ω―é―²―Ä–Η. –£–Α―Ä–Η―Ä–Α–Ι–Κ–Η ―²–Ψ―Ä―¹–Η–Ψ–Ϋ–Ϋ–Η―²–Β ―ä–≥–Μ–Η –Ω–Ψ –Ω–Ψ–Μ–Η–Ω–Β―²–Η–¥–Ϋ–Α―²–Α –≤–Β―Ä–Η–≥–Α –Η –Ω–Ψ ―¹―²―Ä–Α–Ϋ–Η―΅–Ϋ–Η―²–Β –≤–Β―Ä–Η–≥–Η –Ϋ–Α –Α–Φ–Η–Ϋ–Ψ–Κ–Η―¹–Β–Μ–Η–Ϋ–Ϋ–Η―²–Β –Ψ―¹―²–Α―²―ä―Ü–Η –Κ–Ψ–Φ–Ω―é―²―ä―Ä―ä―² –Ψ–Ω―²–Η–Φ–Η–Ζ–Η―Ä–Α –Β–Ϋ–Β―Ä–≥–Η―è―²–Α –≤ ―²―ä―Ä―¹–Β–Ϋ–Β –Ϋ–Α –Φ–Η–Ϋ–Η–Φ―É–Φ. –ù–Ψ –¥–Α ―¹–Β –Ϋ–Α–Φ–Β―Ä–Η –±―ä―Ä–Ζ–Ψ –≥–Μ–Ψ–±–Α–Μ–Ϋ–Η―è –Φ–Η–Ϋ–Η–Φ―É–Φ –Ϋ–Α –Β–¥–Ϋ–Α ―³―É–Ϋ–Κ―Ü–Η―è ―¹ –Φ–Ϋ–Ψ–≥–Ψ –Ω―Ä–Ψ–Φ–Β–Ϋ–Μ–Η–≤–Η –Β –Ϋ–Β–≤―ä–Ζ–Φ–Ψ–Ε–Ϋ–Ψ, –Ω–Ψ―Ä–Α–¥–Η ―²–Ψ–≤–Α ―΅–Β –Η–Φ–Α –Φ–Ϋ–Ψ–Ε–Β―¹―²–Ψ –Μ–Ψ–Κ–Α–Μ–Ϋ–Η –Φ–Η–Ϋ–Η–Φ―É–Φ–Η –Η –Ω―Ä–Ψ–≥―Ä–Α–Φ–Α―²–Α –Ϋ–Β –Φ–Ψ–Ε–Β –¥–Α –Ζ–Ϋ–Α–Β –Κ–Α–Κ –¥–Α –Η–¥–Β –Ω―Ä―è–Κ–Ψ –Κ―ä–Φ βÄû–Ζ–Α–≤–Β―²–Ϋ–Η―èβÄ€ –≥–Μ–Ψ–±–Α–Μ–Β–Ϋ –Φ–Η–Ϋ–Η–Φ―É–Φ. –‰–Ζ―΅–Η―¹–Μ–Β–Ϋ–Ψ –Β, ―΅–Β –Ζ–Α –¥–Α ―¹–Β βÄû–Ω―Ä–Ψ–≥–Ψ–Ϋ―è―²βÄ€ –≤―¹–Η―΅–Κ–Η –≤―ä–Ζ–Φ–Ψ–Ε–Ϋ–Ψ―¹―²–Η –Η ―¹–Β ―¹―²–Η–≥–Ϋ–Β –¥–Ψ –Ψ–Ω―²–Η–Φ–Α–Μ–Ϋ–Α―²–Α ―¹―²―Ä―É–Κ―É―Ä–Α –Ϋ―è–Φ–Α –¥–Α ―¹―²–Η–≥–Ϋ–Β –Η –≤―Ä–Β–Φ–Β―²–Ψ –Ψ―² –Ϋ–Α―΅–Α–Μ–Ψ―²–Ψ –Ϋ–Α –£―¹–Β–Μ–Β–Ϋ–Α―²–Α. –Δ–Ψ–≤–Α –Β ―²–Α–Κ–Α –Ϋ–Α―Ä–Β―΅–Β–Ϋ–Η―è –Ω–Α―Ä–Α–¥–Ψ–Κ―¹ –Ϋ–Α –¦–Β–≤–Η–Ϋ―²–Α–Μ.
–ê –Κ–Α–Κ –Ω―Ä–Η―Ä–Ψ–¥–Α―²–Α ―É―¹–Ω―è–≤–Α –¥–Α –Ϋ–Α–Ω―Ä–Α–≤–Η ―²–Ψ–≤–Α –Ζ–Α –Φ–Η–Μ–Η―¹–Β–Κ―É–Ϋ–¥–Η?! –£ –Ω–Β―Ä–Η–Ψ–¥–Α –Ϋ–Α –±–Η–Ψ–Μ–Ψ–≥–Η―΅–Ϋ–Α―²–Α –Β–≤–Ψ–Μ―é―Ü–Η―è, –Η–Ζ–Ω–Ψ–Μ–Ζ―É–≤–Α–Ι–Κ–Η ―¹–Μ―É―΅–Α–Ι–Ϋ–Η –Φ―É―²–Α―Ü–Η–Η, –±–Β–Μ―²―ä―Ü–Η―²–Β ―¹–Α ―É―¹―ä–≤―ä―Ä―à–Β–Ϋ―¹―²–≤–Α–Μ–Η –Ω―ä―Ä–≤–Η―΅–Ϋ–Α―²–Α ―¹–Η ―¹―²―Ä―É–Κ―²―É―Ä–Α ―²–Α–Κ–Α, ―΅–Β –¥–Α –Ζ–Α–Β–Φ–Α―² ―³―É–Ϋ–Κ―Ü–Η–Ψ–Ϋ–Η―Ä–Α―â–Α –Ω―Ä–Ψ―¹―²―Ä–Α–Ϋ―¹―²–≤–Β–Ϋ–Α ―¹–≥–Μ–Ψ–±–Κ–Α. –Δ–Β ―¹–Α, –Ω–Ψ –¥―É–Φ–Η―²–Β –Ϋ–Α –Η–Ζ–≤–Β―¹―²–Ϋ–Η―è ―²–Β–Ψ―Ä–Β―²–Η–Κ –û. –ë. –ü―²–Η―Ü–Η–Ϋ, –Β–≤–Ψ–Μ―é―Ü–Η–Ψ–Ϋ–Ϋ–Ψ-–Ψ―²―Ä–Β–¥–Α–Κ―²–Η―Ä–Α–Ϋ–Η –±–Η–Ψ–Ω–Ψ–Μ–Η–Φ–Β―Ä–Η.
–û―² –Ϋ–Α―΅–Α–Μ–Ψ –Κ–Ψ–Φ–Ω―é―²―ä―Ä–Ϋ–Η―²–Β –Φ–Β―²–Ψ–¥–Η –Ϋ―è–Φ–Α―² –≤–Η―¹–Ψ–Κ–Α –Ω―Ä–Β–¥―¹–Κ–Α–Ζ–Α―²–Β–Μ–Ϋ–Α ―¹–Ω–Ψ―¹–Ψ–±–Ϋ–Ψ―¹―². –î–Ψ―Ä–Η ―¹–Μ–Β–¥ –≤–Ϋ–Α―¹―è–Ϋ–Β –Ϋ–Α –Η–Ϋ―³–Ψ―Ä–Φ–Α―Ü–Η―è –Ζ–Α ―¹―²―Ä―É–Κ―²―É―Ä–Ϋ–Η―²–Β –Φ–Ψ―²–Η–≤–Η –Ω―Ä–Η –≤–Β―΅–Β –Η–Ζ–≤–Β―¹―²–Ϋ–Η―²–Β ―¹―²―Ä―É–Κ―²―É―Ä–Η, –Ζ–Α –¥–Α ―¹–Β –Ω–Ψ–¥―¹–Κ–Α–Ε–Β –Ω―Ä–Η ―²―ä―Ä―¹–Β–Ϋ–Β –Ϋ–Α –Ϋ–Β–Η–Ζ–≤–Β―¹―²–Ϋ–Α ―¹―²―Ä―É–Κ―²―É―Ä–Α, ―É―¹–Ω–Β―Ö―ä―² –Ϋ–Β –Β –Ω–Ψ–≤–Β―΅–Β –Ψ―² 60%. –Γ –Η–Ζ–Ω–Ψ–Μ–Ζ―É–≤–Α–Ϋ–Β –Ϋ–Α –Β–≤–Ψ–Μ―é―Ü–Η–Ψ–Ϋ–Ϋ–Η ―¹―ä–Ψ–±―Ä–Α–Ε–Β–Ϋ–Η―è –Ω―Ä–Η ―¹―Ä–Α–≤–Ϋ–Β–Ϋ–Η–Β –Ϋ–Α ―¹―Ä–Ψ–¥–Ϋ–Η –±–Β–Μ―²―ä―Ü–Η ―²–Ψ–Ζ–Η –Ω―Ä–Ψ―Ü–Β–Ϋ―² ―¹―²–Η–≥–Α 70.
–ö―ä–Φ 2018 ―². –±–Β–Μ―²―ä―΅–Ϋ–Η―²–Β βÄû–Φ–Ψ–¥–Β–Μ–Η–Β―Ä–ΗβÄ€ –Ζ–Α–Ω–Ψ―΅–≤–Α―² –¥–Α –Ω―Ä–Η–Μ–Α–≥–Α―² –Ω―Ä–Ψ–≥―Ä–Α–Φ–Η –Ψ―¹–Ϋ–Ψ–≤–Α–Ϋ–Η –Ϋ–Α –‰–Ζ–Κ―É―¹―²–≤–Β–Ϋ–Η―è –‰–Ϋ―²–Β–Μ–Β–Κ―² (–‰–‰) –Η –Ϋ–Β–≤―Ä–Ψ–Ϋ–Ϋ–Η –Φ―Ä–Β–Ε–Η. –î―ä―â–Β―Ä–Ϋ–Α―²–Α ―³–Η―Ä–Φ–Α –Ϋ–Α –™―É–≥―ä–Μ βÄ™ DeepMind ―²―Ä–Β–Ϋ–Η―Ä–Α –≤―ä―Ä―Ö―É –Η–Ζ–≤–Β―¹―²–Ϋ–Η –±–Β–Μ―²―ä―΅–Ϋ–Η ―¹―²―Ä―É–Κ―²―É―Ä–Η –Ζ–Α –¥–Α –Η–Ζ–Ω–Ψ–Μ–Ζ–≤–Α ―²–Ψ–≤–Α –Ω―Ä–Η ―²―ä―Ä―¹–Β–Ϋ–Β –Ϋ–Α –Ϋ–Β–Η–Ζ–≤–Β―¹―²–Ϋ–Α ―¹―²―Ä―É–Κ―²―É―Ä–Α, –≤ –Κ–Ψ―è―²–Ψ ―¹–Β ―¹―Ä–Β―â–Α―² ―¹―Ö–Ψ–¥–Ϋ–Η –Ω–Β–Ω―²–Η–¥–Ϋ–Η –Φ–Ψ―²–Η–≤–Η.
–ö―ä–Φ 2020 –≥. AlphaFold2 –Η–Ζ–Ω–Ψ–Μ–Ζ–≤–Α –Φ―Ä–Β–Ε–Α –Ψ―² 182 –Ω―Ä–Ψ―Ü–Β―¹–Ψ―Ä–Α, –Ψ–Ω―²–Η–Φ–Η–Ζ–Η―Ä–Α–Ϋ–Η –Ζ–Α –Φ–Α―à–Η–Ϋ–Ϋ–Ψ –Ψ–±―É―΅–Β–Ϋ–Η–Β, –Η –¥–Ψ―¹―²–Η–≥–Α ―²–Ψ―΅–Ϋ–Ψ―¹―² 92.4% ―¹―Ä–Α–≤–Ϋ–Η–Φ–Α ―¹ –Β–Κ–Ω–Β―Ä–Η–Φ–Β–Ϋ―²–Α–Μ–Ϋ–Η―²–Β ―²–Β―Ö–Ϋ–Η–Κ–Η. –Δ–Α–Ζ–Η –≥–Ψ–¥–Η–Ϋ–Α ―¹ –Η–Ζ–Ω–Ψ–Μ–Ζ–≤–Α–Ϋ–Β –Ϋ–Α –‰–‰ –≥―Ä―É–Ω–Α―²–Α –Ϋ–Α –î–Β–Ι–≤–Η–¥ –ë–Β–Ι–Κ―ä―Ä –Ψ―² –‰–Ϋ―¹―²–Η―²―É―²–Α –Ω–Ψ –Ω―Ä–Ψ–Β–Κ―²–Η―Ä–Α–Ϋ–Β –Ϋ–Α –±–Β–Μ―²―ä―Ü–Η –Ϋ–Α –£–Α―à–Η–Ϋ–≥―²–Ψ–Ϋ―¹–Κ–Η―è –Θ–Ϋ–Η–≤–Β―Ä―¹–Η―²–Β―², ―¹ –Η–Ζ–Ω–Ψ–Μ–Ζ―É–≤–Α–Ϋ–Β –Ϋ–Α ―É―¹―ä–≤―ä―Ä―à–Β–Ϋ―¹―²–≤–Α–Ϋ ―¹–Ψ―³―²―É–Β―Ä RoseTTAFold –Η Alphafold na Deepmind –Ω―Ä–Β–¥―¹–Κ–Α–Ζ–≤–Α―² ―¹―²―Ä―É–Κ―²―É―Ä–Α―²–Α –Ϋ–Α ―Ö–Η–Μ―è–¥–Η –±–Β–Μ―²―ä―Ü–Η. –Γ–Μ–Β–¥ ―²–Ψ–≤–Α –Ω―Ä–Ψ–≤–Β―Ä―è–≤–Α―² ―¹ –Κ–Ψ–Φ–Ω―é―²―ä―Ä–Α –Κ–Ψ–Η –Ψ―² ―²–Β–Ζ–Η –±–Β–Μ―²―ä―Ü–Η –Φ–Ψ–≥–Α―² –¥–Α –Ψ–±―Ä–Α–Ζ―É–≤–Α―² –Κ–Ψ–Φ–Ω–Μ–Β–Κ―¹–Η.
–Δ–Ψ–≤–Α –≤ –Ω–Ψ―¹–Μ–Β–¥―¹―²–≤–Η–Β –Φ–Ψ–Ε–Β –¥–Α ―¹–Β –Ω―Ä–Ψ–≤–Β―Ä―è–≤–Α –Ω–Ψ –Β–Κ–Ω–Β―Ä–Η–Φ–Β–Ϋ―²–Α–Μ–Β–Ϋ –Ω―ä―².
–Γ–Ψ―³―²―É–Β―Ä―ä―² –Ϋ–Α –êlfaFold2 –Η roseTTAFold –Β –Ω―É–±–Μ–Η―΅–Ϋ–Ψ –¥–Ψ―¹―²―ä–Ω–Β–Ϋ –Ζ–Α –≤―¹–Β–Κ–Η, –Κ–Ψ–Ι―²–Ψ –Η―¹–Κ–Α –¥–Α –Ω―Ä–Ψ–Β–Κ―²–Η―Ä–Α –Ϋ–Ψ–≤ –±–Β–Μ―²―ä–Κ –Η–Μ–Η –¥–Α –Ϋ–Α–Φ–Β―Ä–Η ―¹―²―Ä―É–Κ―²―É―Ä–Α―²–Α –Ϋ–Α –±–Β–Μ―²―ä–Κ ―¹ –Η–Ζ–≤–Β―¹―²–Ϋ–Α –Ω―ä―Ä–≤–Η―΅–Ϋ–Α –Ω–Ψ―¹–Μ–Β–¥–Ψ–≤–Α―²–Β–Μ–Ϋ–Ψ―¹―². –ü―Ä–Β–Ζ –Ϋ–Ψ–Β–Φ–≤―Ä–Η ―É―΅–Β–Ϋ–Η –Ψ―² –™–Β―Ä–Φ–Α–Ϋ–Η―è –Η –Γ–ê–© –Η–Ζ–Ω–Ψ–Μ–Ζ―É–≤–Α―² ―²–Ψ–Ζ–Η ―¹–Ψ―³―²―É–Β―Ä,¬†–Κ–Α–Κ―²–Ψ –Η –Κ―Ä–Η–Ψ–Β–Μ–Β–Κ―²―Ä–Ψ–Ϋ–Ϋ–Α –Φ–Η–Κ―Ä–Ψ―¹–Κ–Ψ–Ω–Η―è, –Ζ–Α –¥–Α –Κ–Α―Ä―²–Η―Ä–Α―² ―¹―²―Ä―É–Κ―²―É―Ä–Α―²–Α –Ϋ–Α –Κ–Ψ–Φ–Ω–Μ–Β–Κ―¹, –Ψ–±―Ä–Α–Ζ―É–≤–Α―â –Κ–Μ–Β―²―ä―΅–Ϋ–Α ―è–¥―Ä–Β–Ϋ–Α –Ω–Ψ―Ä–Α. –Δ–Ψ–Ι ―¹–Β ―¹―ä―¹―²–Ψ–Η –Ψ―² 30 ―Ä–Α–Ζ–Μ–Η―΅–Ϋ–Η –±–Β–Μ―²―ä―Ü–Η, –Κ–Ψ–Η―²–Ψ –Κ–Ψ–Ϋ―²―Ä–Ψ–Μ–Η―Ä–Α―² –¥–Ψ―¹―²―ä–Ω–Α –¥–Ψ –Κ–Μ–Β―²―ä―΅–Ϋ–Ψ―²–Ψ ―è–¥―Ä–Ψ. –Γ―ä―â–Η―è―² –Φ–Β―²–Ψ–¥ –Ω–Ψ–Ζ–≤–Ψ–Μ―è–≤–Α –¥–Α ―¹–Β ―²―ä―Ä―¹―è―² –Ϋ–Ψ–≤–Η –Μ–Β–Κ–Α―Ä―¹―²–≤–Α.
 –î–Β–Ι–≤–Η–¥ –ë–Β–Ι–Κ―ä―Ä ―Ä―ä–Κ–Ψ–≤–Ψ–¥–Η –‰–Ϋ―¹―²–Η―²―É―²–Α –Ω–Ψ –ü―Ä–Ψ–Β–Κ―²–Η―Ä–Α–Ϋ–Β –Ϋ–Α –ë–Β–Μ―²―ä―Ü–Η ―¹ –Ϋ–Β–≥–Ψ–≤–Η―²–Β 130 ―¹―ä―²―Ä―É–¥–Ϋ–Η―Ü–Η.
–î–Β–Ι–≤–Η–¥ –ë–Β–Ι–Κ―ä―Ä ―Ä―ä–Κ–Ψ–≤–Ψ–¥–Η –‰–Ϋ―¹―²–Η―²―É―²–Α –Ω–Ψ –ü―Ä–Ψ–Β–Κ―²–Η―Ä–Α–Ϋ–Β –Ϋ–Α –ë–Β–Μ―²―ä―Ü–Η ―¹ –Ϋ–Β–≥–Ψ–≤–Η―²–Β 130 ―¹―ä―²―Ä―É–¥–Ϋ–Η―Ü–Η.
–ö―Ä–Β–¥–Η―²: Ian C. Haydon/IPD
–ö–Α–Κ–≤–Ψ ―â–Β ―¹―²–Α–Ϋ–Β –Α–Κ–Ψ ―¹–Β –Η–Ζ–Ω–Ψ–Μ–Ζ―É–≤–Α –Ψ–±―Ä–Α―²–Β–Ϋ –Ω–Ψ–¥―Ö–Ψ–¥ - –Ψ―² ―²―Ä–Β―²–Η―΅–Ϋ–Α―²–Α (–Ω―Ä–Ψ―¹―²―Ä–Α–Ϋ―¹―²–≤–Β–Ϋ–Α) ―¹―²―Ä―É–Κ―²―É―Ä–Α –Κ―ä–Φ –≤―ä–Ζ–Φ–Ψ–Ε–Ϋ–Η –Ω―ä―Ä–≤–Η―΅–Ϋ–Η ―¹―²―Ä―É–Κ―²―É―Ä–Η, –Κ–Ψ–Η―²–Ψ –¥–Α –¥–Ψ–≤–Β–¥–Α―² –¥–Ψ –Ω–Ψ–¥–Ψ–±–Ϋ–Α ―²―Ä–Η–Φ–Β―Ä–Ϋ–Α –Ψ–Ω–Α–Κ–Ψ–≤–Κ–Α. –Δ–Α–Κ―ä–≤ –Φ–Β―²–Ψ–¥ –Β ―É–¥–Ψ–±–Β–Ϋ –Ζ–Α –Ω―Ä–Ψ–Β–Κ―²–Η―Ä–Α–Ϋ–Β –Ϋ–Α –Ϋ–Ψ–≤–Η –±–Β–Μ―²―ä―Ü–Η.
–Δ–Ψ–≤–Α –Β –¥–Β–Φ–Ψ–Ϋ―¹―²―Ä–Η―Ä–Α–Ϋ–Ψ –≤ –¥–Β–Κ–Β–Φ–≤―Ä–Η–Ι―¹–Κ–Α―²–Α ―¹―²–Α―²–Η―è –Ϋ–Α –‰–≤–Α–Ϋ –ê–Ϋ–Η―à―΅–Β–Ϋ–Κ–Ψ –Η ―¹―ä―²―Ä. –Ψ―² –‰–Ϋ―¹―²–Η―²―É―²–Α –Ϋ–Α –ë–Β–Ι–Κ―ä―Ä.
βÄû–ù–Η–Β –Ζ–Α–Ω–Ψ―΅–Ϋ–Α―Ö–Φ–Β –Ψ―² –Β–¥–Ϋ–Α –Ϋ–Α–Ω―ä–Μ–Ϋ–Ψ ―¹–Μ―É―΅–Α–Ι–Ϋ–Α –Ω–Β–Ω―²–Η–¥–Ϋ–Α –Ω–Ψ―¹–Μ–Β–¥–Ψ–≤–Α―²–Β–Μ–Ϋ–Ψ―¹―² –Η –≤–Ϋ–Α―¹―è―Ö–Φ–Β –Φ―É―²–Α―Ü–Η–Η –¥–Ψ–Κ–Α―²–Ψ –Ϋ–Α―à–Α―²–Α –Ϋ–Β–≤―Ä–Ψ–Ϋ–Ϋ–Α –Φ―Ä–Β–Ε–Α –Ω―Ä–Β–¥―¹–Κ–Α–Ε–Β, ―΅–Β ―²–Β –Φ–Ψ–≥–Α―² –¥–Α ―¹–Β –Ζ–Α–≥―ä–Ϋ–Α―² –≤ ―¹―²–Α–±–Η–Μ–Ϋ–Α ―¹―²―Ä―É–Κ―²―É―Ä–ΑβÄ€ - –Κ–Α–Ζ–≤–Α –ê–Ϋ–Η―à―΅–Β–Ϋ–Κ–Ψ.
 –‰–≤–Α–Ϋ –ê–Ϋ–Η―â―΅–Β–Ϋ–Κ–Ψ, –Ω–Ψ―¹―²–¥–Ψ–Κ –≤ –‰-―²―É―²–Α –Ζ–Α –ë–Β–Μ―²―ä―΅–Ϋ–Ψ –Ω―Ä–Ψ–Β–Κ―²–Η―Ä–Α–Ϋ–Β –≤ –Θ–Ϋ–Η–≤–Β―Ä―¹–Η―²–Β―²–Α –£–Α―à–Η–Ϋ–≥―²–Ψ–Ϋ. –ö―Ä–Β–¥–Η―²: UW IPD Photo
–‰–≤–Α–Ϋ –ê–Ϋ–Η―â―΅–Β–Ϋ–Κ–Ψ, –Ω–Ψ―¹―²–¥–Ψ–Κ –≤ –‰-―²―É―²–Α –Ζ–Α –ë–Β–Μ―²―ä―΅–Ϋ–Ψ –Ω―Ä–Ψ–Β–Κ―²–Η―Ä–Α–Ϋ–Β –≤ –Θ–Ϋ–Η–≤–Β―Ä―¹–Η―²–Β―²–Α –£–Α―à–Η–Ϋ–≥―²–Ψ–Ϋ. –ö―Ä–Β–¥–Η―²: UW IPD Photo
–Δ–Α–Κ―ä–≤ –Ϋ–Α―΅–Η–Ϋ –Ϋ–Α ―²―ä―Ä―¹–Β–Ϋ–Β –Ω―Ä–Η–Μ–Η―΅–Α –Ϋ–Α –Η–≥―Ä–Α―²–Α –Ϋ–Α βÄû―²–Ψ–Ω–Μ–Ψ/―¹―²―É–¥–Β–Ϋ–ΨβÄ€ = βÄû–Ω―Ä–Η–Μ–Η―΅–Α/–Ϋ–Β –Ω―Ä–Η–Μ–Η―΅–ΑβÄ€ –Ϋ–Α ―¹―²―Ä―É–Κ―²―É―Ä–Η―Ä–Α–Ϋ –±–Β–Μ―²―ä–Κ. –‰ –Α–Κ–Ψ –Ω–Ψ–¥–±―Ä–Α–Ϋ–Η―è―² –±–Β–Μ―²―ä–Κ ―¹–Β –Ω―Ä–Ψ–Η–Ζ–≤–Β–¥–Β βÄû–≤ –Β–Ω―Ä―É–≤–Β―²–Κ–ΑβÄ€, –Β–Κ―¹–Ω–Β―Ä–Η–Φ–Β–Ϋ―²–Α–Μ–Ϋ–Ψ ―â–Β –Φ–Ψ–Ε–Β –¥–Α ―¹–Β –Β –Ω–Ψ–¥―²–≤―ä―Ä–¥–Η ―Ä–Β–Ζ―É–Μ―²–Α―²―ä―² –Ψ―² –Φ–Ψ–¥–Β–Μ–Η―Ä–Α–Ϋ–Β―²–Ψ. –Δ–Α–Κ―ä–≤ –Ω–Ψ–¥―Ö–Ψ–¥ –Η–Ζ―¹–Μ–Β–¥–Ψ–≤–Α―²–Β–Μ―¹–Κ–Η―è―² ―²–Η–Φ –Ϋ–Α―Ä–Η―΅–Α βÄû―Ö–Α–Μ―é―Ü–Η–Ϋ–Α―Ü–Η―èβÄ€ –Ϋ–Α –±–Β–Μ―²―ä―Ü–Η.
–Δ–Β―Ä–Φ–Η–Ϋ―ä―² "―Ö–Α–Μ―é―Ü–Η–Ϋ–Α―Ü–Η―è" ―¹–Β –Η–Ζ–Ω–Ψ–Μ–Ζ–≤–Α –≤―ä–≤ –≤―Ä―ä–Ζ–Κ–Α ―¹ ―Ä–Α–Ζ―Ä–Α–±–Ψ―²–Κ–Α –Ϋ–Α –‰–‰ ―²–Η–Φ–Α –Ϋ–Α –™―É–≥―ä–Μ. –½–Α–Ω–Ψ―΅–≤–Α–Ι–Κ–Η –Ψ―² –Ψ–±―Ä–Α–Ζ ―¹―ä―¹ ―¹–Μ―É―΅–Α–Ι–Ϋ–Η (―Ä–Α–Ϋ–¥–Ψ–Φ–Η–Ζ–Η―Ä–Α–Ϋ–Η) –Ω–Η–Κ―¹–Β–Μ–Η –Ϋ–Β–≤―Ä–Ψ–Ϋ–Ϋ–Η―²–Β –Φ―Ä–Β–Ε–Η, ―²―Ä–Β–Ϋ–Η―Ä–Α–Ϋ–Η –¥–Α ―Ä–Α–Ζ–Ω–Ψ–Ζ–Ϋ–Α–≤–Α―² –Ϋ–Α–Ω―Ä–Η–Φ–Β―Ä –Ω–Μ–Ψ–¥, ―¹–Β βÄû–Ζ–Α–Ω–Η―²–≤–Α―²βÄ€ –¥–Ψ–Κ–Ψ–Μ–Κ–Ψ –Ψ–±―Ä–Α–Ζ―ä―² –Ω―Ä–Η–Μ–Η―΅–Α –Ϋ–Α –±–Α–Ϋ–Α–Ϋ? –Γ–Μ–Β–¥ –Ω–Ψ―Ä–Β–¥–Ϋ–Η –Ω―Ä–Ψ–±–≤–Α–Ϋ–Η―è –≤―ä–Ω―Ä–Ψ―¹―ä―² ―¹–Β –Ζ–Α–¥–Α–≤–Α –Ψ―²–Ϋ–Ψ–≤–Ψ –Η –Ψ―²–Ϋ–Ψ–≤–Ψ, –≤―¹―è–Κ–Α –Ω―Ä–Ψ–Φ―è–Ϋ–Α ―¹–Β –Ζ–Α–Ω–Α–Ζ–≤–Α, ―¹―²–Η–≥–Α –¥–Α –Ζ–Α―¹–Η–Μ–≤–Α βÄû–±–Α–Ϋ–Α–Ϋ–Ψ-–Ω–Ψ–¥–Ψ–±–Ϋ–Η―²–ΒβÄ€ ―Ö–Α―Ä–Α–Κ―²–Β―Ä–Η―¹―²–Η–Κ–Η. –ü―Ä–Ψ―Ü–Β―¹―ä―² ―¹–Β –Ω–Ψ–≤―²–Α―Ä―è ―Ö–Η–Μ―è–¥–Ψ–Κ―Ä–Α―²–Ϋ–Ψ. –ö―Ä–Α–Ι–Ϋ–Η―è―² ―Ä–Β–Ζ―É–Μ―²–Α―² –Β ―è–≤–Ϋ–Ψ –±–Α–Ϋ–Α–Ϋ–Ψ-–Ω–Ψ–¥–Ψ–±–Β–Ϋ, –Ϋ–Ψ –Ω―Ä–Η–Μ–Η―΅–Α –Ω–Ψ–≤–Β―΅–Β –Ϋ–Α –Κ–Α―Ä―²–Η–Ϋ–Α –≤ ―¹―²–Η–Μ –Α–Μ–Α –Κ―É–±–Η–Ζ―ä–Φ –Η–Μ–Η –Η–Φ–Ω―Ä–Β―¹–Η–Ψ–Ϋ–Η–Ζ―ä–Φ, ―¹―è–Κ–Α―à ―Ö―É–¥–Ψ–Ε–Ϋ–Η–Κ―ä―² –Β –Ω―Ä–Η–Β–Φ–Α–Μ ―Ö–Α–Μ―é―Ü–Η–Ϋ–Ψ–≥–Β–Ϋ. (–Ω–Ψ –î–Ε–Ψ–Ϋ –Δ–Η–Φ―ä―Ä - Getting software to βÄ€hallucinateβÄù reasonable protein structures, ArsTechnica)
 –Γ–Μ―É―΅–Α–Β–Ϋ ―à―É–Φ(–≤–Μ―è–≤–Ψ) –Ω―Ä–Β–Φ–Η–Ϋ–Α–≤–Α –≤ –±–Α–Ϋ–Α–Ϋ–Ψ–Ω–Ψ–¥–Ψ–±–Ϋ–Η ―Ö–Α–Μ―é―Ü–Η–Ϋ–Α―Ü–Η―è(–≤–¥―è―¹–Ϋ–Ψ) ―΅―Ä–Β–Ζ –Ω–Ψ–≤―²–Ψ―Ä–Ϋ–Η ―²―ä―Ä―¹–Β–Ϋ–Β―²–Α ―¹ –±–Α–Ϋ–Α–Ϋ–Ψ―Ä–Α–Ζ–Ω–Ψ–Ζ–Ϋ–Α–≤–Α―â–Η―è –Η–Ζ–Κ―É―¹―²–≤–Β–Ϋ –Η–Ϋ―²–Β–Μ–Β–Κ―² (–‰–‰).
–Γ–Μ―É―΅–Α–Β–Ϋ ―à―É–Φ(–≤–Μ―è–≤–Ψ) –Ω―Ä–Β–Φ–Η–Ϋ–Α–≤–Α –≤ –±–Α–Ϋ–Α–Ϋ–Ψ–Ω–Ψ–¥–Ψ–±–Ϋ–Η ―Ö–Α–Μ―é―Ü–Η–Ϋ–Α―Ü–Η―è(–≤–¥―è―¹–Ϋ–Ψ) ―΅―Ä–Β–Ζ –Ω–Ψ–≤―²–Ψ―Ä–Ϋ–Η ―²―ä―Ä―¹–Β–Ϋ–Β―²–Α ―¹ –±–Α–Ϋ–Α–Ϋ–Ψ―Ä–Α–Ζ–Ω–Ψ–Ζ–Ϋ–Α–≤–Α―â–Η―è –Η–Ζ–Κ―É―¹―²–≤–Β–Ϋ –Η–Ϋ―²–Β–Μ–Β–Κ―² (–‰–‰).
–ö―Ä–Β–¥–Η―²: ¬†photo Google
–Γ–Ψ―³―²―É–Β―Ä―ä―² ―Ö–Α–Μ―é―Ü–Η–Ϋ–Η―Ä–Α –Ω―Ä–Η–Β–Φ–Μ–Η–≤–Η –±–Β–Μ―²―ä―΅–Ϋ–Η ―¹―²―Ä―É–Κ―²―É―Ä–Η. –‰―²–Β―Ä–Α―²–Η–≤–Ϋ–Ψ –Ω―Ä–Ψ–≤–Β―Ä―è–≤–Α –¥–Α–Μ–Η βÄû―²–Ψ–≤–Α –Ω―Ä–Η–Μ–Η―΅–Α –Ϋ–Α –±–Β–Μ―²―ä–ΚβÄ€. –£ –Κ–Ψ–Φ–Ω―é―²―ä―Ä–Ϋ–Η―è –Φ–Ψ–¥–Β–Μ ―΅―Ä–Β–Ζ –Φ―É―²–Η―Ä–Α–Ϋ–Β –Ϋ–Α ―¹–Μ―É―΅–Α–Ι–Ϋ–Ψ –Α–Φ–Η–Ϋ–Ψ–Κ–Η―¹–Β–Μ–Η–Ϋ–Η –Ω–Ψ –¥―ä–Μ–Ε–Η–Ϋ–Α―²–Α –Ϋ–Α –Ω–Ψ–Μ–Η–Ω–Β–Ω―²–Η–¥–Ϋ–Α―²–Α –≤–Β―Ä–Η–≥–Α ―¹–Β ―¹–Μ–Β–¥–Η –¥–Α–Μ–Η ―²–Ψ–≤–Α –≤–Ψ–¥–Η –¥–Ψ –Ω–Ψ-―¹―²―Ä―É–Κ―²―É―Ä–Η―Ä–Α–Ϋ –±–Β–Μ―²―ä–Κ. –Δ–Ψ–≥–Α–≤–Α ―¹–Β –Ω―Ä–Α–≤–Η ―¹–Μ–Β–¥–≤–Α―â–Α –Φ―É―²–Α―Ü–Η―è –Η ―²–Α–Κ–Α –≤ 20 ―Ö–Η–Μ―è–¥–Η –Η―²–Β―Ä–Α―²–Η–≤–Ϋ–Η ―¹―²―ä–Ω–Κ–Η. –£ ―Ä–Β–Ζ―É–Μ―²–Α―² ―É―΅–Β–Ϋ–Η―²–Β –Ϋ–Α–Φ–Η―Ä–Α―² 129 –Ϋ–Ψ–≤–Η –±–Β–Μ―²―ä―Ü–Η, ―¹―ä―¹ ―¹―²―Ä―É–Κ―²―É―Ä–Η –Ω–Ψ–¥–Ψ–±–Ϋ–Η –Ϋ–Α –±–Β–Μ―²―ä―Ü–Η, ―¹―Ä–Β―â–Α–Ϋ–Η –≤ –Ω―Ä–Η―Ä–Ψ–¥–Α―²–Α. –½–Α –¥–Α ―¹–Β –Ω―Ä–Ψ–≤–Β―Ä―è―² ―²–Α–Κ–Α –Φ–Ψ–¥–Β–Μ–Η―Ä–Α–Ϋ–Η―²–Β –Ϋ–Ψ–≤–Η ―¹―²―Ä―É–Κ―²―É―Ä–Η, ―É―΅–Β–Ϋ–Η―²–Β ―¹–Η–Ϋ―²–Β–Ζ–Η―Ä–Α―² ―²–Β–Ζ–Η –±–Β–Μ―²―ä―Ü–Η –≤ ―΅―Ä–Β–≤–Ϋ–Α―²–Α –±–Α–Κ―²–Β―Ä–Η―è –ï―à–Β―Ä–Η―Ö–Η―è –ö–Ψ–Μ–Η –≤ –Μ–Α–±–Ψ―Ä–Α―²–Ψ―Ä–Η―è –Η –Ϋ–Α–Φ–Η―Ä–Α―², ―΅–Β 27 –Ψ―² ―²―è―Ö ―¹―ä–≤–Ω–Α–¥–Α―² –Ω–Ψ –Ω―Ä–Ψ―¹―²―Ä–Α–Ϋ―¹―²–≤–Β–Ϋ–Α ―¹―²―Ä―É–Κ―²―É―Ä–Α ―¹ –Ω―Ä–Β–¥―¹–Κ–Α–Ζ–Α–Ϋ–Η―²–Β ―¹―²―Ä―É–Κ―²―É―Ä–Η.
–ï―²–Ψ –Κ–Α–Κ –Η–Ζ–≥–Μ–Β–Ε–¥–Α –Β–¥–Ϋ–Α –Ψ―² –Ϋ–Ψ–≤–Ψ–Ω―Ä–Ψ–Β–Κ―²–Η―Ä–Α–Ϋ–Η―²–Β ―Ö–Α–Μ―é―Ü–Η–Ϋ–Η―Ä–Α–Ϋ–Η –Ψ―² ―¹–Ψ―³―²―É–Β―Ä–Α ―¹―²―Ä―É–Κ―²―É―Ä–Η, ―¹―Ä–Α–≤–Ϋ–Β–Ϋ–Α ―¹ –Β–Κ―¹–Ω–Β―Ä–Η–Φ–Β–Ϋ―²–Α–Μ–Ϋ–Η―è –Κ―Ä–Η―¹―²–Α–Μ–Ψ–≥―Ä–Α―³―¹–Κ–Η ―Ä–Β–Ζ―É–Μ―²–Α―².
 –Γ―Ä–Α–≤–Ϋ–Β–Ϋ–Η–Β –Ϋ–Α βÄû―Ö–Α–Μ―é―Ü–Η–Ϋ–Η―Ä–Α–Ϋ–ΑβÄ€ ―¹―²―Ä―É–Κ―²―É―Ä–Α (–≤ ―¹–Η–Ϋ―¨–Ψ) ―¹–Ω―Ä―è–Φ–Ψ –Κ―Ä–Η―¹―²–Α–Μ–Ϋ–Α―²–Α (–≤ ―¹–Η–≤–Ψ). –£–¥―è―¹–Ϋ–Ψ –¥–Ψ–Μ―É ―¹–Α –Ω–Ψ–Κ–Α–Ζ–Α–Ϋ–Η –Η ―¹―²―Ä–Α–Ϋ–Η―΅–Ϋ–Η―²–Β –Α–Φ–Η–Ϋ–Ψ–Κ–Η―¹–Β–Μ–Η–Ϋ–Ϋ–Η –≤–Β―Ä–Η–≥–Η. –Λ–Η–≥. –Ω–Ψ Anishchenko, I. –Η ―¹―ä―²―Ä.
–Γ―Ä–Α–≤–Ϋ–Β–Ϋ–Η–Β –Ϋ–Α βÄû―Ö–Α–Μ―é―Ü–Η–Ϋ–Η―Ä–Α–Ϋ–ΑβÄ€ ―¹―²―Ä―É–Κ―²―É―Ä–Α (–≤ ―¹–Η–Ϋ―¨–Ψ) ―¹–Ω―Ä―è–Φ–Ψ –Κ―Ä–Η―¹―²–Α–Μ–Ϋ–Α―²–Α (–≤ ―¹–Η–≤–Ψ). –£–¥―è―¹–Ϋ–Ψ –¥–Ψ–Μ―É ―¹–Α –Ω–Ψ–Κ–Α–Ζ–Α–Ϋ–Η –Η ―¹―²―Ä–Α–Ϋ–Η―΅–Ϋ–Η―²–Β –Α–Φ–Η–Ϋ–Ψ–Κ–Η―¹–Β–Μ–Η–Ϋ–Ϋ–Η –≤–Β―Ä–Η–≥–Η. –Λ–Η–≥. –Ω–Ψ Anishchenko, I. –Η ―¹―ä―²―Ä.
–ö–Ψ–Φ–±–Η–Ϋ–Η―Ä–Α–Ϋ–Β―²–Ψ –Ϋ–Α –Ω―Ä–Β–¥―¹–Κ–Α–Ζ–≤–Α–Ϋ–Β –Ϋ–Α –±–Β–Μ―²―ä―΅–Ϋ–Α―²–Α ―¹―²―Ä―É―²―É―Ä–Α ―¹ –¥―ä–Μ–±–Ψ–Κ–Ψ –Ψ–±―É―΅–Β–Ϋ–Η–Β ―¹ –Φ–Β―²–Ψ–¥–Η―²–Β –Ϋ–Α –‰–‰ –Ψ―²–Κ―Ä–Η–≤–Α –Ϋ–Ψ–≤–Η –≤―ä–Ζ–Φ–Ψ–Ε–Ϋ–Ψ―¹―²–Η –Ω―Ä–Η –Ω―Ä–Ψ–Β–Κ―²–Η―Ä–Α–Ϋ–Β―²–Ψ –Ϋ–Α –±–Β–Μ―²―ä―Ü–Η. –ï―²–Ψ –Κ–Α–Κ–≤–Ψ –Κ–Α–Ζ–≤–Α –î–Β–Ι–≤–Η–¥ –ë–Β–Ι–Κ―ä―Ä, –Κ–Ψ–Ι―²–Ψ –Ω–Ψ–Μ―É―΅–Α–≤–Α –Ϋ–Α–≥―Ä–Α–¥–Α―²–Α –Ϋ–Α ―¹–Ω. –Γ–Α–Ι―ä–Ϋ―¹ –Ζ–Α –≤–Ψ–¥–Β―â–Ψ –Ψ―²–Κ―Ä–Η―²–Η–Β:
βÄ€–‰–Ζ–Ω–Ψ–Μ–Ζ―É–≤–Α–Ϋ–Β―²–Ψ –Ϋ–Α –Ζ–Α–¥―ä–Μ–±–Ψ―΅–Β–Ϋ–Ψ –Ψ–±―É―΅–Β–Ϋ–Η–Β (deep learning) –Ϋ–Α –Ϋ–Β–≤―Ä–Ψ–Ϋ–Ϋ–Η―²–Β –Φ―Ä–Β–Ε–Η, –Κ–Ψ–Η―²–Ψ ―¹–Α ―¹–Β ―¹–Α–Φ–Ψ―²―Ä–Β–Ϋ–Η―Ä–Α–Μ–Η –≤―ä―Ä―Ö―É –Ψ–±―â–Η―²–Β ―¹―²―Ä―É–Κ―²―É―Ä–Ϋ–Η –Ω―Ä–Η–Ϋ―Ü–Η–Ω–Η –Ζ–Α –Ω–Ψ―¹―²―Ä–Ψ―è–≤–Α–Ϋ–Β –Ϋ–Α –±–Β–Μ―²―ä―΅–Ϋ–Η ―¹―²―Ä―É–Κ―²―É―Ä–Η –¥–Α–≤–Α –≤―ä–Ζ–Φ–Ψ–Ε–Ϋ–Ψ―¹―² –¥–Α ―¹–Β ―³–Ψ–Κ―É―¹–Η―Ä–Α–Φ–Β –Ω―Ä―è–Κ–Ψ –≤―ä―Ä―Ö―É ―³―É–Ϋ–Κ―Ü–Η–Ψ–Ϋ–Α–Μ–Ϋ–Η―²–Β ―΅–Α―¹―²–Η –Ϋ–Α –Ϋ–Ψ–≤–Η –±–Β–Μ―²―ä―Ü–Η ―¹ –Ψ–≥–Μ–Β–¥ –Ϋ–Α ―¹–Ω–Β―Ü–Η―³–Η―΅–Ϋ–Η –Ω―Ä–Η–Μ–Ψ–Ε–Β–Ϋ–Η―è.βÄ€
 Credit: Alecsandra Dragoi for Nature
Credit: Alecsandra Dragoi for Nature
–Γ–Ω–Η―¹–Α–Ϋ–Η–Β―²–Ψ Nature ―¹―ä―â–Ψ –Η–Φ–Α –Κ–Μ–Α―¹–Α―Ü–Η―è βÄ™ –Ϋ–Α –Ϋ–Α–Ι-–Ζ–Α―¹–Μ―É–Ε–Η–Μ–Η―²–Β ―É―΅–Β–Ϋ–Η¬†–Ω―Ä–Β–Ζ –≥–Ψ–¥–Η–Ϋ–Α―²–Α. –£ –¥–Β–Κ–Β–Φ–≤―Ä–Η–Ι―¹–Κ–Η―è –±―Ä–Ψ–Ι –Ϋ–Α ―¹–Ω–Η―¹–Α–Ϋ–Η–Β―²–Ψ –Β–¥–Η–Ϋ –Ψ―² ―²―è―Ö
–Β –î–Ε–Ψ–Ϋ –î–Ε―ä–Φ–Ω―ä―Ä (John Jumper).¬† –Δ–Ψ–Ι ―Ä―ä–Κ–Ψ–≤–Ψ–¥–Η ―²–Η–Φ–Α –≤ –Κ–Ψ–Φ–Ω–Α–Ϋ–Η―è―²–Α DeepMind,¬†―¹―ä–Ζ–¥–Α–Μ–Α –Η–Ϋ―¹―²―Ä―É–Φ–Β–Ϋ―²–Α AlphaFold2, –Κ–Ψ–Ι―²–Ψ –Η–Ζ–Ω–Ψ–Μ–Ζ–Α –‰–‰ –Φ–Β―²–Ψ–¥–Η –Η¬†–Ϋ–Β–≤―Ä–Ψ–Ϋ–Ϋ–Η –Φ―Ä–Β–Ε–Η, –Ζ–Α –Κ–Ψ–Η―²–Ψ ―¹–Ω–Ψ–Φ–Β–Ϋ–Α―Ö–Φ–Β –Ω–Ψ-–≥–Ψ―Ä–Β, –Η –Ω―Ä–Β–¥―¹–Κ–Α–Ζ–≤–Α¬†―¹―²―Ä―É–Κ―²―É―Ä–Α―²–Α –Ϋ–Α –±–Β–Μ―²―ä―Ü–Η ―¹ ―³–Β–Ϋ–Ψ–Φ–Β–Ϋ–Α–Μ–Ϋ–Α ―²–Ψ―΅–Ϋ–Ψ―¹―², –Ω―Ä–Β–≤―ä–Ζ―Ö–Ψ–Ε–¥–Α–Ι–Κ–Η¬†–≤―¹–Η―΅–Κ–Η –Ω–Ψ–¥–Ψ–±–Ϋ–Η –Κ–Ψ–Ϋ–Κ―É―Ä–Β―²–Ϋ–Η –Ω–Ψ–¥―Ö–Ψ–¥–Η. –ü―Ä–Β–Ζ ―é–Μ–Η¬†–î–Ε―ä–Φ–Ω―ä―Ä –Ψ–±–Ϋ–Α―Ä–Ψ–¥–≤–Α ―¹–Ψ―³―²―É–Β―Ä–Α ―¹–Η –Η –Ζ–Α–Β–¥–Ϋ–Ψ ―¹ –ï–≤―Ä–Ψ–Ω–Β–Ι―¹–Κ–Α―²–Α –Μ–Α–±–Ψ―Ä–Α―²–Ψ―Ä–Η―è¬†–Ω–Ψ –Φ–Ψ–Μ–Β–Κ―É–Μ―è―Ä–Ϋ–Α –±–Η–Ψ–Μ–Ψ–≥–Η―è –Ω―Ä–Β–¥―¹–Κ–Α–Ζ–≤–Α―² ―¹―²―Ä―É–Κ―²―É―Ä–Η―²–Β –Ϋ–Α –≤―¹–Η―΅–Κ–Η¬†–±–Β–Μ―²―ä―Ü–Η,¬† ―¹―Ä–Β―â–Α–Ϋ–Η –Ω―Ä–Η ―Ö–Ψ―Ä–Α―²–Α –Η 20 –¥―Ä―É–≥–Η –Φ–Ψ–¥–Β–Μ–Ϋ–Η –Ψ―Ä–≥–Α–Ϋ–Η–Ζ–Φ–Η -¬†–Ψ–±―â–Ψ 250000.
–ü–Μ–Α–Ϋ―ä―² –Η–Φ –Β –¥–Ψ –Κ―Ä–Α―è –Ϋ–Α –Η–¥–Ϋ–Α―²–Α –≥–Ψ–¥–Η–Ϋ–Α –¥–Α¬†–Ω―Ä–Β–¥―¹–Κ–Α–Ε–Α―² ―¹―²―Ä―É–Κ―²―É―Ä–Α―²–Α –Ϋ–Α –Ω–Ψ–Μ–Ψ–≤–Η–Ϋ–Α―²–Α –Ψ―² –≤―¹–Η―΅–Κ–Η –Η–Ζ–≤–Β―¹―²–Ϋ–Η¬†–±–Β–Μ―²―ä―Ü–Η, –Κ–Ψ–Η―²–Ψ ―¹–Α –Ψ–Κ–Ψ–Μ–Ψ 130 –Φ–Η–Μ–Η–Ψ–Ϋ–Α. –Δ–Ψ–≤–Α ―â–Β –Β ―²―ä―Ä–Ε–Β―¹―²–≤–Ψ –Ϋ–Α ―΅–Ψ–≤–Β―à–Κ–Η―è –Η¬†–Η–Ζ–Κ―É―¹―²–≤–Β–Ϋ–Η―è –Η–Ϋ―²–Β–Μ–Β–Κ―². –ü―Ä–Ψ–±–Μ–Β–Φ―ä―² ―¹―ä―¹ –Ω―Ä–Β–¥―¹–Κ–Α–Ζ–≤–Α–Ϋ–Β –Ϋ–Α¬†―¹―²―Ä―É–Κ―²―É―Ä–Α―²–Α –Ϋ–Α –±–Β–Μ―²―ä―Ü–Η―²–Β –≤–Β―΅–Β –Ϋ–Α –Ω―Ä–Α–Κ―²–Η–Κ–Α –Ϋ–Β ―¹―ä―â–Β―¹―²–≤―É–≤–Α.
–£―ä―Ä―Ö–Ψ–≤–Ψ –Ω–Ψ―¹―²–Η–Ε–Β–Ϋ–Η–Β ―¹ –Ω–Ψ–Φ–Ψ―â―²–Α –Ϋ–Α AlphaFold2 e –Ϋ–Α–Φ–Η―Ä–Α–Ϋ–Β –Ϋ–Α¬†―¹―²―Ä―É–Κ―²―É―Ä–Α―²–Α –Ϋ–Α ―è–¥―Ä–Β–Ϋ–Α―²–Α –Ω–Ψ―Ä–Α –Ϋ–Α –Β―É–Κ–Α―Ä–Η–Ψ―²–Ϋ–Α―²–Α –Κ–Μ–Β―²–Κ–Α. –Δ―è ―¹–Β ―¹―ä―¹―²–Ψ–Η¬†–Ψ―² 30-―²–Η–Ϋ–Α ―Ä–Α–Ζ–Μ–Η―΅–Ϋ–Η –±–Β–Μ―²―ä―Ü–Η –≤ ―¹–Μ–Ψ–Ε–Β–Ϋ –Κ–Ψ–Φ–Ω–Μ–Β–Κ―¹ –Ψ–±―â–Ψ –Ϋ–Α ―Ö–Η–Μ―è–¥–Α¬†–±–Β–Μ―²―ä―΅–Ϋ–Η –≤–Β―Ä–Η–≥–Η –Κ–Μ–Β―²–Κ–Α.
βÄ€–Δ–Α–Κ–Α ―¹–Ψ―³―²―É–Β―Ä―ä―² –Ω―Ä–Ψ–Φ–Β–Ϋ―è ―Ä–Α–±–Ψ―²–Α―²–Α –Ϋ–Α¬†–Β–Κ―¹–Ω–Β―Ä–Η–Φ–Β–Ϋ―²–Α―²–Ψ―Ä–Η―²–Β, ―²–Ψ–≤–Α –Β –Ζ–Α–±–Β–Μ–Β–Ε–Η―²–Β–Μ–Β–Ϋ ―É―¹–Ω–Β―Ö",¬†–Κ–Α–Ζ–≤–Α –î–Ε―ä–Φ–Ω―ä―Ä.
(–Ω–Ψ News Feature Nature 15 December 2021 Nature βÄôs 10: ten people who helped shape science in
2021 )
***
–‰–Ζ–Ω–Ψ–Μ–Ζ–≤–Α–Ϋ–Α –Μ–Η―²–Β―Ä–Α―²―É―Ä–Α: Anishchenko, I., Pellock, S.J., Chidyausiku, T.M.¬†et al.¬†De novo protein design by deep network hallucination.¬†Nature¬†600,¬†547βÄ™552 (2021). https://doi.org/10.1038/s41586-021-04184-w
–ê–≤―²–Ψ―Ä―ä―² –ö–Ψ–Ϋ―¹―²–Α–Ϋ―²–Η–Ϋ –ß–Η–Ω–Β–≤ –Β –Φ–Ψ–Μ–Β–Κ―É–Μ―è―Ä–Β–Ϋ –Η –Κ–Μ–Β―²―ä―΅–Β–Ϋ –±–Η–Ψ–Μ–Ψ–≥ –Ψ―² –Θ–Ϋ–Η–≤–Β―Ä―¹–Η―²–Β―²–Α –Γ―²–Ψ–Ϋ–Η –ë―Ä―É–Κ –≤ –ù―é –ô–Ψ―Ä–Κ.
–û―â–Β –Ω–Ψ ―²–Β–Φ–Α―²–Α
–•–Η–≤–Ψ―²―ä―²
–™–Β–Ϋ–Ϋ–Η ―²–Β―Ä–Α–Ω–Η–Η –Ζ–Α –Μ–Β–Κ―É–≤–Α–Ϋ–Β –Ϋ–Α ―¹―ä―Ä–Ω–Ψ–≤–Η–¥–Ϋ–Ψ-–Κ–Μ–Β―²―ä―΅–Ϋ–Α –Α–Ϋ–Β–Φ–Η―è –Η –±–Β―²–Α-―²–Α–Μ–Α―¹–Β–Φ–Η―è
–€–Β–¥–Η―Ü–Η–Ϋ–Α
–™–Β–Ϋ–Ϋ–Α ―²–Β―Ä–Α–Ω–Η―è ―¹ CRISPR ―΅―Ä–Β–Ζ ―²―Ä–Α–Ϋ―¹―³―É–Ζ–Η―è –Ϋ–Α –†–ù–ö –≤ –Κ―Ä―ä–≤―²–Α

–•–Η–≤–Ψ―²―ä―²
–‰–Ζ–Κ―É―¹―²–≤–Β–Ϋ –Η–Ϋ―²–Β–Μ–Β–Κ―² βÄû–Ω―Ä–Β–¥―¹–Κ–Α–Ζ–≤–Α ―¹–Μ–Β–¥–≤–Α―â–Η―è –Κ–Ψ―Ä–Ψ–Ϋ–Α–≤–Η―Ä―É―¹βÄ€















–ö–Ψ–Φ–Β–Ϋ―²–Α―Ä–Η
–€–Ψ–Μ―è, ―Ä–Β–≥–Η―¹―²―Ä–Η―Ä–Α–Ι―²–Β ―¹–Β –Ψ―² T–Θ–ö!
–ê–Κ–Ψ –≤–Β―΅–Β –Η–Φ–Α―²–Β ―Ä–Β–≥–Η―¹―²―Ä–Α―Ü–Η―è, –Ϋ–Α―²–Η―¹–Ϋ–Β―²–Β –Δ–Θ–ö!
–ù―è–Φ–Α –Κ–Ψ–Φ–Β–Ϋ―²–Α―Ä–Η –Κ―ä–Φ ―²–Α–Ζ–Η –Ϋ–Ψ–≤–Η–Ϋ–Α !
–ü–Ψ―¹–Μ–Β–¥–Ϋ–Η –Κ–Ψ–Φ–Β–Ϋ―²–Α―Ä–Η
Gunteer
–ü―Ä–Β―¹―²–Η–Ε–Ϋ–Α –Ϋ–Α–≥―Ä–Α–¥–Α –Ψ―² –ë–ê–ù ―¹–Ω–Β―΅–Β–Μ–Η –≥–Μ–Α–≤–Ϋ–Η―è―² ―Ä–Β–¥–Α–Κ―²–Ψ―Ä –Ϋ–Α –ù–Α―É–Κ–ΑOFFNews
–Ξ―Ä–Η―¹―²–Η―è–Ϋ–Η–Ϋ
–Δ–Ψ–≤–Α –Β –Κ―Ä–Α–Μ―è―² –Ϋ–Α ―²–Η–Κ–≤–Η―²–Β: –Δ–Η–Κ–≤–Α―²–Α –Φ―É ―²–Β–Ε–Η –Ϋ–Α–¥ –Β–¥–Η–Ϋ –Β–¥–Η–Ϋ ―²–Ψ–Ϋ
dolivo
–Γ–Α―Ö–Α―Ä–Α –Ψ―΅–Α–Κ–≤–Α 75% ―É–≤–Β–Μ–Η―΅–Β–Ϋ–Η–Β –Ϋ–Α –≤–Α–Μ–Β–Ε–Η―²–Β –¥–Ψ 2100 –≥.
dolivo
–½–Β–Φ–Ϋ–Α―²–Α ―è–±―ä–Μ–Κ–Α: ―¹―²–Α―Ä–Α –Κ―É–Μ―²―É―Ä–Α –Ζ–Α –Ϋ–Ψ–≤–Η―²–Β –Κ–Μ–Η–Φ–Α―²–Η―΅–Ϋ–Η –≤―Ä–Β–Φ–Β–Ϋ–Α