

–С—А–Є—В–∞–љ—Б–Ї–∞—В–∞ –Ї–Њ–Љ–њ–∞–љ–Є—П –Ј–∞ –Є–Ј–Ї—Г—Б—В–≤–µ–љ –Є–љ—В–µ–ї–µ–Ї—В DeepMind –µ —А–∞–Ј–≥–∞–і–∞–ї–∞ —Б—В—А—Г–Ї—В—Г—А–∞—В–∞ –љ–∞ –њ–Њ—З—В–Є –≤—Б–Є—З–Ї–Є –Є–Ј–≤–µ—Б—В–љ–Є –љ–∞ –љ–∞—Г–Ї–∞—В–∞ –њ—А–Њ—В–µ–Є–љ–Є.
–Ш–Ј—Б–ї–µ–і–Њ–≤–∞—В–µ–ї–Є—В–µ —Б–∞ –њ–Њ—Б—В–Є–≥–љ–∞–ї–Є —В–Њ–Ј–Є —Г—Б–њ–µ—Е —Б –њ–Њ–Љ–Њ—Й—В–∞ –љ–∞ –њ—А–Њ–≥—А–∞–Љ–∞—В–∞ AlphaFold, –Ї–Њ—П—В–Њ DeepMind —А–∞–Ј—А–∞–±–Њ—В–≤–∞ –Ј–∞ –њ—К—А–≤–Є –њ—К—В –њ—А–µ–Ј 2018 –≥. –Є –њ—Г—Б–Ї–∞ –њ—Г–±–ї–Є—З–љ–Њ –њ—А–µ–Ј —О–ї–Є 2021 –≥. –Я—А–Њ–≥—А–∞–Љ–∞—В–∞ —Б –Њ—В–≤–Њ—А–µ–љ –Ї–Њ–і –Љ–Њ–ґ–µ –і–∞ –њ—А–µ–і—Б–Ї–∞–ґ–µ 3D —Б—В—А—Г–Ї—В—Г—А–∞—В–∞ –љ–∞ –і–∞–і–µ–љ –њ—А–Њ—В–µ–Є–љ –Њ—В –њ–Њ—Б–ї–µ–і–Њ–≤–∞—В–µ–ї–љ–Њ—Б—В—В–∞ –љ–∞ –∞–Љ–Є–љ–Њ–Ї–Є—Б–µ–ї–Є–љ–Є—В–µ - –≥—А–∞–і–Є–≤–љ–Є—В–µ –µ–ї–µ–Љ–µ–љ—В–Є, —Б—К—Б—В–∞–≤—П—Й–Є –њ—А–Њ—В–µ–Є–љ–Є—В–µ. –°—В—А—Г–Ї—В—Г—А–∞—В–∞ –љ–∞ –њ—А–Њ—В–µ–Є–љ–∞ –і–Є–Ї—В—Г–≤–∞ –љ–µ–≥–Њ–≤–Є—В–µ —Д—Г–љ–Ї—Ж–Є–Є, —В–∞–Ї–∞ —З–µ –±–∞–Ј–∞—В–∞ –і–∞–љ–љ–Є –Њ—В 200 –Љ–Є–ї–Є–Њ–љ–∞ –њ—А–Њ—В–µ–Є–љ–Њ–≤–Є —Б—В—А—Г–Ї—В—Г—А–Є, –Є–і–µ–љ—В–Є—Д–Є—Ж–Є—А–∞–љ–Є –Њ—В AlphaFold, –Є–Љ–∞ –њ–Њ—В–µ–љ—Ж–Є–∞–ї–∞ –і–∞ –њ–Њ–Љ–Њ–≥–љ–µ –Ј–∞ –Є–і–µ–љ—В–Є—Д–Є—Ж–Є—А–∞–љ–µ—В–Њ –љ–∞ –љ–Њ–≤–Є –њ—А–Њ—В–µ–Є–љ–Њ–≤–Є –Љ–∞—И–Є–љ–Є, –Њ—В –Ї–Њ–Є—В–Њ —Е–Њ—А–∞—В–∞ –Љ–Њ–≥–∞—В –і–∞ —Б–µ –≤—К–Ј–њ–Њ–ї–Ј–≤–∞—В.
–Я—А–Њ–≥—А–∞–Љ–∞—В–∞ AlphaFold –Є–Ј–њ–Њ–ї–Ј–≤–∞ –љ–µ–≤—А–Њ–љ–љ–Є –Љ—А–µ–ґ–Є –Ј–∞ –Є–Ј–≤—К—А—И–≤–∞–љ–µ –љ–∞ –і—К–ї–±–Њ–Ї–Њ —Б–∞–Љ–Њ–Њ–±—Г—З–µ–љ–Є–µ, –Є–і–µ–љ—В–Є—Д–Є—Ж–Є—А–∞–є–Ї–Є –Љ–Њ–і–µ–ї–Є –≤ –њ–Њ—Б–ї–µ–і–Њ–≤–∞—В–µ–ї–љ–Њ—Б—В–Є –Є —Б—В—А—Г–Ї—В—Г—А–Є –љ–∞ –њ—А–Њ—В–µ–Є–љ–Є, –Њ—В–Ї—А–Є—В–Є –≤ –≥–ї–Њ–±–∞–ї–љ–∞ –±–∞–Ј–∞ –і–∞–љ–љ–Є. –Ґ—К–є –Ї–∞—В–Њ —Б–µ –Њ–±—Г—З–∞–≤–∞ —Б —В–µ—З–µ–љ–Є–µ –љ–∞ –≤—А–µ–Љ–µ—В–Њ, –њ—А–Њ–≥—А–∞–Љ–∞—В–∞ –Љ–Њ–ґ–µ –і–∞ –Є–і–µ–љ—В–Є—Д–Є—Ж–Є—А–∞ —Б—В—А—Г–Ї—В—Г—А–∞—В–∞ –љ–∞ –њ—А–Њ—В–µ–Є–љ–∞ –Ј–∞ –Љ–Є–љ—Г—В–Є. –Ъ–∞–Ї —А–∞–±–Њ—В–Є –і—К–ї–±–Њ–Ї–Њ—В–Њ —Б–∞–Љ–Њ–Њ–±—Г—З–µ–љ–Є–µ –љ–∞ –љ–µ–≤—А–Њ–љ–љ–Є—В–µ –Љ—А–µ–ґ–Є –Љ–Њ–ґ–µ –і–∞ —А–∞–Ј–±–µ—А–µ—В–µ –≤ —В–∞–Ј–Є —Б—В–∞—В–Є—П –љ–∞ –Э–∞—Г–Ї–∞OFFNews.
–С–∞–Ј–∞—В–∞ –і–∞–љ–љ–Є –Љ–Њ–ґ–µ –і–∞ –≤–Ї–ї—О—З–≤–∞ –љ–∞–њ—А–Є–Љ–µ—А –њ—А–Њ—В–µ–Є–љ–Є, –Ї–Њ–Є—В–Њ –Љ–Њ–≥–∞—В –і–∞ –њ–Њ–Љ–Њ–≥–љ–∞—В –Ј–∞ —А–µ—Ж–Є–Ї–ї–Є—А–∞–љ–µ—В–Њ –љ–∞ –њ–ї–∞—Б—В–Љ–∞—Б–Є, —Б—К–Њ–±—Й–∞–≤–∞¬†The Guardian.
"–С–µ—И–µ –љ–Є –љ–µ–Њ–±—Е–Њ–і–Є–Љ–Њ –і–Њ—Б—В–∞ –і—К–ї–≥–Њ –≤—А–µ–Љ–µ, –Ј–∞ –і–∞ –њ—А–µ–≥–ї–µ–і–∞–Љ–µ —В–∞–Ј–Є –Њ–≥—А–Њ–Љ–љ–∞ –±–∞–Ј–∞ –і–∞–љ–љ–Є –Њ—В —Б—В—А—Г–Ї—В—Г—А–Є, –љ–Њ [—В–Њ–≤–∞] –љ–Є —А–∞–Ј–Ї—А–Є —Ж—П–ї –љ–∞–±–Њ—А –Њ—В –љ–Њ–≤–Є —В—А–Є–Є–Ј–Љ–µ—А–љ–Є —Д–Њ—А–Љ–Є, –Ї–Њ–Є—В–Њ –љ–µ –±—П—Е–Љ–µ –≤–Є–ґ–і–∞–ї–Є –і–Њ—Б–µ–≥–∞ –Є –Ї–Њ–Є—В–Њ –і–µ–є—Б—В–≤–Є—В–µ–ї–љ–Њ –Љ–Њ–≥–∞—В –і–∞ —А–∞–Ј–≥—А–∞–ґ–і–∞—В –њ–ї–∞—Б—В–Љ–∞—Б–Є", —А–∞–Ј–Ї–∞–Ј–≤–∞ –њ—А–µ–і The Guardian –Ф–ґ–Њ–љ –Ь–∞–Ї–У–Є–є–љ (John McGeehan), –њ—А–Њ—Д–µ—Б–Њ—А –њ–Њ —Б—В—А—Г–Ї—В—Г—А–љ–∞ –±–Є–Њ–ї–Њ–≥–Є—П –≤ –£–љ–Є–≤–µ—А—Б–Є—В–µ—В–∞ –≤ –Я–Њ—А—В—Б–Љ—Г—В, –Т–µ–ї–Є–Ї–Њ–±—А–Є—В–∞–љ–Є—П. "–Э–∞–ї–Є—Ж–µ –µ –њ—К–ї–љ–∞ –њ—А–Њ–Љ—П–љ–∞ –љ–∞ –њ–∞—А–∞–і–Є–≥–Љ–∞—В–∞. –Ь–Њ–ґ–µ–Љ –љ–∞–Є—Б—В–Є–љ–∞ –і–∞ —Г—Б–Ї–Њ—А–Є–Љ –љ–∞–њ—А–µ–і—К–Ї–∞ —Б–Є - –Є —В–Њ–≤–∞ –љ–Є –њ–Њ–Љ–∞–≥–∞ –і–∞ –љ–∞—Б–Њ—З–Є–Љ —В–µ–Ј–Є —Ж–µ–љ–љ–Є —А–µ—Б—Г—А—Б–Є –Ї—К–Љ –Ј–љ–∞—З–Є–Љ–Є—В–µ –љ–µ—Й–∞."

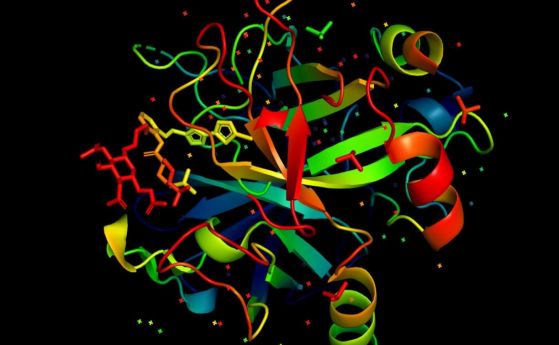
AlphaFold –љ–∞ DeepMind —Б—К–Ј–і–∞–≤–∞ 3D –Є–Ј–Њ–±—А–∞–ґ–µ–љ–Є—П –љ–∞ –њ—А–Њ—В–µ–Є–љ–Њ–≤–Є —Б—В—А—Г–Ї—В—Г—А–Є.¬†–Ъ—А–µ–і–Є—В: Deepmind
–Я—А–Њ—В–µ–Є–љ–Є—В–µ —Б–∞ –Ї–∞—В–Њ –Љ–∞–ї–Ї–Є, –љ–µ—А–∞–Ј–≥–∞–і–∞–µ–Љ–Є –њ—К–Ј–µ–ї–Є. –Ґ–µ —Б–µ –њ—А–Њ–Є–Ј–≤–µ–ґ–і–∞—В –Њ—В —А–∞–Ј–ї–Є—З–љ–Є –Њ—А–≥–∞–љ–Є–Ј–Љ–Є - –Њ—В –±–∞–Ї—В–µ—А–Є–Є –і–Њ —А–∞—Б—В–µ–љ–Є—П –Є –ґ–Є–≤–Њ—В–љ–Є, –Є –Ї–Њ–≥–∞—В–Њ —Б–µ —Б—К–Ј–і–∞–і–∞—В, —Б–µ —Б–≥—К–≤–∞—В –Ј–∞ –Љ–Є–ї–Є—Б–µ–Ї—Г–љ–і–Є, –љ–Њ —Б—В—А—Г–Ї—В—Г—А–Є—В–µ –Є–Љ —Б–∞ —В–Њ–ї–Ї–Њ–≤–∞ —Б–ї–Њ–ґ–љ–Є, —З–µ –Њ–њ–Є—В–Є—В–µ –і–∞ —Б–µ –њ–Њ–Ј–љ–∞–µ –Ї–∞–Ї–≤–∞ —Д–Њ—А–Љ–∞ —Й–µ –њ—А–Є–µ–Љ–∞—В —Б–∞ –њ–Њ—З—В–Є –љ–µ–≤—К–Ј–Љ–Њ–ґ–љ–Є.
–Т¬†—Б—В–∞—В–Є—П –Њ—В 1969 –≥. –°–∞–є—А—К—Б –Ы–µ–≤–Є–љ—В–∞–ї (Cyrus Levinthal), –∞–Љ–µ—А–Є–Ї–∞–љ—Б–Ї–Є –Љ–Њ–ї–µ–Ї—Г–ї—П—А–µ–љ –±–Є–Њ–ї–Њ–≥, –њ–Њ—Б–Њ—З–≤–∞ –њ–∞—А–∞–і–Њ–Ї—Б–∞, —З–µ –њ—А–Њ—В–µ–Є–љ–Є—В–µ —Б–µ —Б–≥—К–≤–∞—В —В–Њ–ї–Ї–Њ–≤–∞ –±—К—А–Ј–Њ –Є —В–Њ—З–љ–Њ, –≤—К–њ—А–µ–Ї–Є —З–µ –Є–Љ–∞—В –Њ–≥—А–Њ–Љ–µ–љ –±—А–Њ–є –≤—К–Ј–Љ–Њ–ґ–љ–Є –Ї–Њ–љ—Д–Є–≥—Г—А–∞—Ж–Є–Є, –Є–Ј—З–Є—Б–ї—П–≤–∞–є–Ї–Є, —З–µ –і–∞–і–µ–љ –њ—А–Њ—В–µ–Є–љ –Љ–Њ–ґ–µ –і–∞ –Є–Љ–∞ 10300 –≤—К–Ј–Љ–Њ–ґ–љ–Є –Ї—А–∞–є–љ–Є —Д–Њ—А–Љ–Є.
–Я–Њ —В–Њ–Ј–Є –љ–∞—З–Є–љ, –њ–Є—И–µ –Ы–µ–≤–Є–љ—В–∞–ї, –∞–Ї–Њ —З–Њ–≤–µ–Ї —Б–µ –Њ–њ–Є—В–∞ –і–∞ —Б—В–Є–≥–љ–µ –і–Њ –њ—А–∞–≤–Є–ї–љ–∞—В–∞ —Д–Њ—А–Љ–∞ –љ–∞ –±–µ–ї—В—К–Ї–∞, –Ї–∞—В–Њ –Є–Ј–њ—А–Њ–±–≤–∞ –≤—Б—П–Ї–∞ –Ї–Њ–љ—Д–Є–≥—Г—А–∞—Ж–Є—П –µ–і–љ–∞ –њ–Њ –µ–і–љ–∞, —Й–µ –Љ—Г –µ –љ–µ–Њ–±—Е–Њ–і–Є–Љ–Њ –њ–Њ–≤–µ—З–µ –≤—А–µ–Љ–µ, –Њ—В–Ї–Њ–ї–Ї–Њ—В–Њ –Т—Б–µ–ї–µ–љ–∞—В–∞ –µ —Б—К—Й–µ—Б—В–≤—Г–≤–∞–ї–∞ –і–Њ—Б–µ–≥–∞, –Ј–∞ –і–∞ —Б—В–Є–≥–љ–µ –і–Њ –њ—А–∞–≤–Є–ї–љ–Є—П –Њ—В–≥–Њ–≤–Њ—А.
–£—З–µ–љ–Є—В–µ —А–∞–Ј–њ–Њ–ї–∞–≥–∞—В —Б –љ–∞—З–Є–љ–Є –і–∞ –≤–Є–Ј—Г–∞–ї–Є–Ј–Є—А–∞—В –њ—А–Њ—В–µ–Є–љ–Є—В–µ –Є –і–∞ –∞–љ–∞–ї–Є–Ј–Є—А–∞—В —В–µ—Е–љ–Є—В–µ —Б—В—А—Г–Ї—В—Г—А–Є, –љ–Њ —В–Њ–≤–∞ –µ –±–∞–≤–љ–∞ –Є —В—А—Г–і–љ–∞ —А–∞–±–Њ—В–∞. –Э–∞–є-—А–∞–Ј–њ—А–Њ—Б—В—А–∞–љ–µ–љ–Є—П—В –љ–∞—З–Є–љ –Ј–∞ –≤–Є–Ј—Г–∞–ї–Є–Ј–Є—А–∞–љ–µ –љ–∞ –њ—А–Њ—В–µ–Є–љ–Є –µ —А–µ–љ—В–≥–µ–љ–Њ–≤–∞—В–∞ –Ї—А–Є—Б—В–∞–ї–Њ–≥—А–∞—Д–Є—П, –Ї–∞–Ї—В–Њ —Б–µ –њ–Њ—Б–Њ—З–≤–∞ –≤ —Б–њ–Є—Б–∞–љ–Є–µ Nature, –Ї–Њ—П—В–Њ –≤–Ї–ї—О—З–≤–∞ –Є–Ј–њ—А–∞—Й–∞–љ–µ –љ–∞ —А–µ–љ—В–≥–µ–љ–Њ–≤–Є –ї—К—З–Є –Ї—К–Љ —В–≤—К—А–і–Є –Ї—А–Є—Б—В–∞–ї–Є –љ–∞ –њ—А–Њ—В–µ–Є–љ–Є –Є –Є–Ј–Љ–µ—А–≤–∞–љ–µ –љ–∞ –љ–∞—З–Є–љ–∞, –њ–Њ –Ї–Њ–є—В–Њ —В–µ–Ј–Є –ї—К—З–Є —Б–µ —А–∞–Ј—Б–µ–є–≤–∞—В, –Ј–∞ –і–∞ —Б–µ –Њ–њ—А–µ–і–µ–ї–Є –Ї–∞–Ї –µ —Г—Б—В—А–Њ–µ–љ –њ—А–Њ—В–µ–Є–љ—К—В. –Ґ–∞–Ј–Є –µ–Ї—Б–њ–µ—А–Є–Љ–µ–љ—В–∞–ї–љ–∞ —А–∞–±–Њ—В–∞ –µ —Г—Б—В–∞–љ–Њ–≤–Є–ї–∞ —Д–Њ—А–Љ–∞—В–∞ –љ–∞ –Њ–Ї–Њ–ї–Њ 190 000 –њ—А–Њ—В–µ–Є–љ–∞, —Б—К–Њ–±—Й–∞–≤–∞ DeepMind.
–Ь–Є–љ–∞–ї–∞—В–∞ –≥–Њ–і–Є–љ–∞ DeepMind –њ—Г–±–ї–Є–Ї—Г–≤–∞ –њ—А–Њ–≥–љ–Њ–Ј–Є –Ј–∞ —Д–Њ—А–Љ–∞—В–∞ –љ–∞ –≤—Б–µ–Ї–Є –њ—А–Њ—В–µ–Є–љ –≤ —З–Њ–≤–µ—И–Ї–Њ—В–Њ —В—П–ї–Њ –Є –Ј–∞ 20 –≤–Є–і–∞ –ґ–Є–≤–Њ—В–љ–Є, –Ї–Њ–Є—В–Њ —Б–∞ –Њ–±–µ–Ї—В –љ–∞ –Є–Ј—Б–ї–µ–і–≤–∞–љ–µ. –°–µ–≥–∞ —В–µ —Б–∞ —А–∞–Ј—И–Є—А–Є–ї–Є —В–µ–Ј–Є –њ—А–Њ–≥–љ–Њ–Ј–Є –і–Њ –њ—А–Њ—В–µ–Є–љ–Є –≤ –њ–Њ—З—В–Є –≤—Б–Є—З–Ї–Њ.
"–Ґ–∞–Ј–Є –∞–Ї—В—Г–∞–ї–Є–Ј–∞—Ж–Є—П –≤–Ї–ї—О—З–≤–∞ –њ—А–µ–і—Б–Ї–∞–Ј–∞–љ–Є —Б—В—А—Г–Ї—В—Г—А–Є –Ј–∞ —А–∞—Б—В–µ–љ–Є—П, –±–∞–Ї—В–µ—А–Є–Є, –ґ–Є–≤–Њ—В–љ–Є –Є –і—А—Г–≥–Є –Њ—А–≥–∞–љ–Є–Ј–Љ–Є, –Ї–Њ–µ—В–Њ –Њ—В–Ї—А–Є–≤–∞ –Љ–љ–Њ–≥–Њ –љ–Њ–≤–Є –≤—К–Ј–Љ–Њ–ґ–љ–Њ—Б—В–Є –Ј–∞ –Є–Ј—Б–ї–µ–і–Њ–≤–∞—В–µ–ї–Є—В–µ –і–∞ –Є–Ј–њ–Њ–ї–Ј–≤–∞—В AlphaFold, –Ј–∞ –і–∞ –љ–∞–њ—А–µ–і–љ–∞—В –≤ —А–∞–±–Њ—В–∞—В–∞ —Б–Є –њ–Њ –≤–∞–ґ–љ–Є –≤—К–њ—А–Њ—Б–Є, –≤–Ї–ї—О—З–Є—В–µ–ї–љ–Њ —Г—Б—В–Њ–є—З–Є–≤–Њ—Б—В, –њ—А–Њ–і–Њ–≤–Њ–ї—Б—В–≤–µ–љ–∞ –љ–µ—Б–Є–≥—Г—А–љ–Њ—Б—В –Є –њ—А–µ–љ–µ–±—А–µ–≥–≤–∞–љ–Є –±–Њ–ї–µ—Б—В–Є", –Ј–∞—П–≤—П–≤–∞—В –њ—А–µ–і—Б—В–∞–≤–Є—В–µ–ї–Є –љ–∞ DeepMind.
–Ф–∞ –љ–∞–Ї–∞—А–∞–Љ–µ –њ—А–Њ—В–µ–Є–љ–Є—В–µ –і–∞ —А–∞–±–Њ—В—П—В
AlphaFold —А–∞–±–Њ—В–Є, –љ–∞—В—А—Г–њ–≤–∞–є–Ї–Є –Ј–љ–∞–љ–Є—П –Ј–∞ –∞–Љ–Є–љ–Њ–Ї–Є—Б–µ–ї–Є–љ–љ–Є—В–µ –њ–Њ—Б–ї–µ–і–Њ–≤–∞—В–µ–ї–љ–Њ—Б—В–Є –Є –≤–Ј–∞–Є–Љ–Њ–і–µ–є—Б—В–≤–Є—П, –і–Њ–Ї–∞—В–Њ —Б–µ –Њ–њ–Є—В–≤–∞ –і–∞ –Є–љ—В–µ—А–њ—А–µ—В–Є—А–∞ –њ—А–Њ—В–µ–Є–љ–Њ–≤–Є—В–µ —Б—В—А—Г–Ї—В—Г—А–Є. –°–µ–≥–∞ –∞–ї–≥–Њ—А–Є—В—К–Љ—К—В –Љ–Њ–ґ–µ –і–∞ –њ—А–µ–і—Б–Ї–∞–Ј–≤–∞ —Д–Њ—А–Љ–Є—В–µ –љ–∞ –њ—А–Њ—В–µ–Є–љ–Є—В–µ –Ј–∞ –Љ–Є–љ—Г—В–Є —Б —В–Њ—З–љ–Њ—Б—В –і–Њ –∞—В–Њ–Љ.
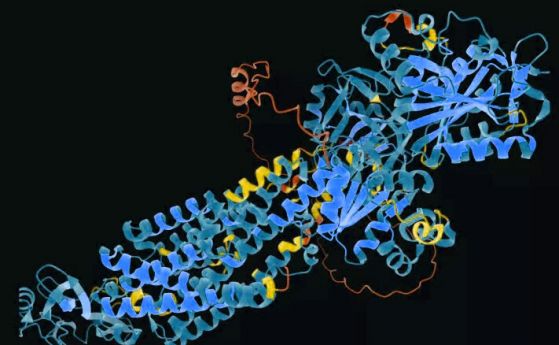
 AlphaFold –њ—А–Њ–≥–љ–Њ–Ј–Є—А–∞ –Љ–љ–Њ–≥–Њ —В–Њ—З–љ–Њ —Б—В—А—Г–Ї—В—Г—А–Є, —Б—А–∞–≤–љ—П–≤–∞–љ–Є —Б –µ–Ї—Б–њ–µ—А–Є–Љ–µ–љ—В–∞–ї–љ–Є —А–µ–Ј—Г–ї—В–∞—В–Є. –Ъ—А–µ–і–Є—В: DeepMind
AlphaFold –њ—А–Њ–≥–љ–Њ–Ј–Є—А–∞ –Љ–љ–Њ–≥–Њ —В–Њ—З–љ–Њ —Б—В—А—Г–Ї—В—Г—А–Є, —Б—А–∞–≤–љ—П–≤–∞–љ–Є —Б –µ–Ї—Б–њ–µ—А–Є–Љ–µ–љ—В–∞–ї–љ–Є —А–µ–Ј—Г–ї—В–∞—В–Є. –Ъ—А–µ–і–Є—В: DeepMind
–Ш–Ј—Б–ї–µ–і–Њ–≤–∞—В–µ–ї–Є—В–µ –≤–µ—З–µ –Є–Ј–њ–Њ–ї–Ј–≤–∞—В –њ–ї–Њ–і–Њ–≤–µ—В–µ –љ–∞ —В—А—Г–і–∞ –љ–∞ AlphaFold. –°–њ–Њ—А–µ–і The Guardian –њ—А–Њ–≥—А–∞–Љ–∞—В–∞ –µ –њ–Њ–Ј–≤–Њ–ї–Є–ї–∞ –љ–∞ –Є–Ј—Б–ї–µ–і–Њ–≤–∞—В–µ–ї–Є—В–µ –љ–∞–є-–љ–∞–Ї—А–∞—П –і–∞ —Е–∞—А–∞–Ї—В–µ—А–Є–Ј–Є—А–∞—В –Ї–ї—О—З–Њ–≤ –њ—А–Њ—В–µ–Є–љ –љ–∞ –Љ–∞–ї–∞—А–Є–є–љ–Є—П –њ–∞—А–∞–Ј–Є—В, –Ї–Њ–є—В–Њ –і–Њ—Б–µ–≥–∞ –±–µ –љ–µ–і–Њ—Б—В—К–њ–µ–љ –Ј–∞ —А–µ–љ—В–≥–µ–љ–Њ–≤–∞ –Ї—А–Є—Б—В–∞–ї–Њ–≥—А–∞—Д–Є—П. –°–њ–Њ—А–µ–і –Є–Ј—Б–ї–µ–і–Њ–≤–∞—В–µ–ї–Є—В–µ —В–Њ–≤–∞ –Љ–Њ–ґ–µ –і–∞ –њ–Њ–і–Њ–±—А–Є —А–∞–Ј—А–∞–±–Њ—В–≤–∞–љ–µ—В–Њ –љ–∞ –≤–∞–Ї—Б–Є–љ–Є —Б—А–µ—Й—Г –±–Њ–ї–µ—Б—В—В–∞.
–Т –Э–Њ—А–≤–µ–ґ–Ї–Є—П —Г–љ–Є–≤–µ—А—Б–Є—В–µ—В –њ–Њ –љ–∞—Г–Ї–Є –Ј–∞ –ґ–Є–≤–Њ—В–∞ –Є–Ј—Б–ї–µ–і–Њ–≤–∞—В–µ–ї—П—В –љ–∞ –Љ–µ–і–Њ–љ–Њ—Б–љ–Є—В–µ –њ—З–µ–ї–Є –Т–Є–ї–і–µ –Ы–µ–є–њ–∞—А—В (Vilde Leipart) –Є–Ј–њ–Њ–ї–Ј–≤–∞ AlphaFold, –Ј–∞ –і–∞ —А–∞–Ј–Ї—А–Є–µ —Б—В—А—Г–Ї—В—Г—А–∞—В–∞ –љ–∞ –≤–Є—В–µ–ї–Њ–≥–µ–љ–Є–љ–∞ - —А–µ–њ—А–Њ–і—Г–Ї—В–Є–≤–µ–љ –Є –Є–Љ—Г–љ–µ–љ –њ—А–Њ—В–µ–Є–љ, –Ї–Њ–є—В–Њ —Б–µ –њ—А–Њ–Є–Ј–≤–µ–ґ–і–∞ –Њ—В –≤—Б–Є—З–Ї–Є –ґ–Є–≤–Њ—В–љ–Є, —Б–љ–∞—Б—П—Й–Є —П–є—Ж–∞. –Ю—В–Ї—А–Є—В–Є–µ—В–Њ –Љ–Њ–ґ–µ –і–∞ –і–Њ–≤–µ–і–µ –і–Њ –љ–Њ–≤–Є –љ–∞—З–Є–љ–Є –Ј–∞ –Ј–∞—Й–Є—В–∞ –љ–∞ –≤–∞–ґ–љ–Є –ґ–Є–≤–Њ—В–љ–Є, –Ї–Њ–Є—В–Њ —Б–љ–∞—Б—П—В —П–є—Ж–∞, –Ї–∞—В–Њ –Љ–µ–і–Њ–љ–Њ—Б–љ–Є—В–µ –њ—З–µ–ї–Є –Є —А–Є–±–Є—В–µ, –Њ—В –±–Њ–ї–µ—Б—В–Є, –њ–Є—И–µ –Ы–µ–є–њ–∞—А—В –≤ –њ—Г–±–ї–Є–Ї–∞—Ж–Є—П –≤ –±–ї–Њ–≥–∞ –љ–∞ DeepMind.
–Р–љ—В–Є–±–Є–Њ—В–Є—З–љ–∞—В–∞ —А–µ–Ј–Є—Б—В–µ–љ—В–љ–Њ—Б—В, –њ—А–Є –Ї–Њ—П—В–Њ –±–∞–Ї—В–µ—А–Є–Є—В–µ —А–∞–Ј–≤–Є–≤–∞—В —Б–њ–Њ—Б–Њ–±–љ–Њ—Б—В—В–∞ –і–∞ –њ–Њ–±–µ–ґ–і–∞–≤–∞—В —В–µ–Ј–Є –ї–µ–Ї–∞—А—Б—В–≤–∞, –±—К—А–Ј–Њ —Б–µ –њ—А–µ–≤—А—К—Й–∞ –≤ –љ–∞—А–∞—Б—В–≤–∞—Й –≥–ї–Њ–±–∞–ї–µ–љ –њ—А–Њ–±–ї–µ–Љ. –° –љ–Њ–≤–Є—В–µ –∞–љ—В–Є–±–Є–Њ—В–Є—Ж–Є, —Г—З–µ–љ–Є –Ї–∞—В–Њ –Ь–∞—А—Б–µ–ї–Њ –°–Њ—Г—Б–∞ (Marcelo Sousa) –Є –Ь–µ–≥–∞–љ –Ь–Є—З—К–ї (Megan Mitchell) –Њ—В –£–љ–Є–≤–µ—А—Б–Є—В–µ—В–∞ –≤ –Ъ–Њ–ї–Њ—А–∞–і–Њ –С–Њ—Г–ї–і—К—А —В—К—А—Б—П—В —А–∞–Ј–ї–Є—З–µ–љ –њ–Њ–і—Е–Њ–і: –Ї–∞—В–Њ —Б–µ –љ–∞—Б–Њ—З–≤–∞—В –Ї—К–Љ —Б–∞–Љ–Є—П –Љ–µ—Е–∞–љ–Є–Ј—К–Љ –љ–∞ —А–µ–Ј–Є—Б—В–µ–љ—В–љ–Њ—Б—В. –С–ї–Њ–Ї–Є—А–∞–љ–µ—В–Њ –љ–∞ –Љ–µ—Е–∞–љ–Є–Ј–Љ–∞ –љ–∞ —А–µ–Ј–Є—Б—В–µ–љ—В–љ–Њ—Б—В, –Ї–Њ–љ—В—А–Њ–ї–Є—А–∞–љ –Њ—В –µ–љ–Ј–Є–Љ–Є, —В—А—П–±–≤–∞ –і–∞ –њ–Њ–Ј–≤–Њ–ї–Є –љ–∞ —Б—К—Й–µ—Б—В–≤—Г–≤–∞—Й–Є—В–µ –∞–љ—В–Є–±–Є–Њ—В–Є—Ж–Є –і–∞ –њ—А–Њ–і—К–ї–ґ–∞—В –і–∞ —Д—Г–љ–Ї—Ж–Є–Њ–љ–Є—А–∞—В. –Э–Њ —А–∞–Ј–±–Є—А–∞–љ–µ—В–Њ –љ–∞ —Б—В—А—Г–Ї—В—Г—А–∞—В–∞ –љ–∞ —В–µ–Ј–Є –µ–љ–Ј–Є–Љ–Є —Б–µ –Њ–Ї–∞–Ј–∞ –љ–µ–≤–µ—А–Њ—П—В–љ–Њ –њ—А–µ–і–Є–Ј–≤–Є–Ї–∞—В–µ–ї—Б—В–≤–Њ —Б —З–Є—Б—В–Њ –µ–Ї—Б–њ–µ—А–Є–Љ–µ–љ—В–∞–ї–љ–Є –Љ–µ—В–Њ–і–Є вАУ –і–Њ–Ї–∞—В–Њ –µ–Ї–Є–њ—К—В –љ–µ —Б–µ –љ–∞—В—К–Ї–≤–∞ –љ–∞ AlphaFold. –Ґ–µ —Б–њ–Њ–і–µ–ї—П—В, —З–µ —Б–∞ –≤–і—К—Е–љ–Њ–≤–µ–љ–Є¬†–Њ—В¬†–њ–Њ—В–µ–љ—Ж–Є–∞–ї–∞ –љ–∞ —В–µ–Ј–Є –і–∞–љ–љ–Є –Є –Ї–∞–Ї–≤–Њ –Љ–Њ–ґ–µ –і–∞ –Њ–Ј–љ–∞—З–∞–≤–∞ —В–Њ–≤–∞ –Ј–∞ –∞–љ—В–Є–±–Є–Њ—В–Є—З–љ–∞—В–∞ —А–µ–Ј–Є—Б—В–µ–љ—В–љ–Њ—Б—В.
–Я—А–Њ–≥—А–∞–Љ–∞—В–∞ —Б—К—Й–Њ —В–∞–Ї–∞ –і–∞–≤–∞ –Є–љ—Д–Њ—А–Љ–∞—Ж–Є—П –Ј–∞ —В—К—А—Б–µ–љ–µ—В–Њ –љ–∞ –љ–Њ–≤–Є —Д–∞—А–Љ–∞—Ж–µ–≤—В–Є—З–љ–Є –њ—А–Њ–і—Г–Ї—В–Є, –Њ—В–±–µ–ї—П–Ј–≤–∞ –†–Њ–Ј–∞–љ–∞ –Ъ–∞–њ–µ–ї–µ—А (Rosana Kapeller), –≥–ї–∞–≤–µ–љ –Є–Ј–њ—К–ї–љ–Є—В–µ–ї–µ–љ –і–Є—А–µ–Ї—В–Њ—А –љ–∞ ROME Therapeutics, –≤ –Є–Ј—П–≤–ї–µ–љ–Є–µ—В–Њ –љ–∞ DeepMind.
"–°–Ї–Њ—А–Њ—Б—В—В–∞ –Є —В–Њ—З–љ–Њ—Б—В—В–∞ –љ–∞ AlphaFold —Г—Б–Ї–Њ—А—П–≤–∞—В –њ—А–Њ—Ж–µ—Б–∞ –љ–∞ –Њ—В–Ї—А–Є–≤–∞–љ–µ –љ–∞ –ї–µ–Ї–∞—А—Б—В–≤–∞", –њ–Њ–і—З–µ—А—В–∞–≤–∞ –Ъ–∞–њ–µ–ї–µ—А,¬†"–Є –љ–Є–µ —Б–Љ–µ –µ–і–≤–∞ –≤ –љ–∞—З–∞–ї–Њ—В–Њ –љ–∞ –Њ—Б—К–Ј–љ–∞–≤–∞–љ–µ—В–Њ –љ–∞ –љ–µ–є–љ–Њ—В–Њ –≤—К–Ј–і–µ–є—Б—В–≤–Є–µ –≤—К—А—Е—Г –њ–Њ-–±—К—А–Ј–Њ—В–Њ –і–Њ—Б—В–Є–≥–∞–љ–µ –љ–∞ –љ–Њ–≤–Є –ї–µ–Ї–∞—А—Б—В–≤–∞ –і–Њ –њ–∞—Ж–Є–µ–љ—В–Є—В–µ".
–°–њ—А–∞–≤–Ї–∞:¬†Structure of the malaria vaccine candidate Pfs48/45 and its recognition by transmission blocking antibodies
Kuang-Ting Ko, Frank Lennartz, David Mekhaiel, Bora Guloglu, Arianna Marini, Danielle J. Deuker, Carole A. Long, Matthijs M. Jore, Kazutoyo Miura, Sumi Biswas, View ORCID ProfileMatthew K. Higgins
doi: https://doi.org/10.1101/2022.05.24.493318 
–Ш–Ј—В–Њ—З–љ–Є–Ї:¬†DeepMind AI has discovered the structure of nearly every protein known to science,¬†Stephanie Pappas,¬†Live Science.
–Ю—Й–µ –њ–Њ —В–µ–Љ–∞—В–∞

–Ц–Є–≤–Њ—В—К—В
–Я–Њ—Б—В–Є–ґ–µ–љ–Є—П—В–∞ –≤ –љ–∞—Г–Ї–∞—В–∞ –Ј–∞ 2021 –≥. —Б–њ–Њ—А–µ–і —Б–њ–Є—Б–∞–љ–Є–µ Science
–Ц–Є–≤–Њ—В—К—В
–Ш–Ј–Ї—Г—Б—В–≤–µ–љ –Є–љ—В–µ–ї–µ–Ї—В –µ —А–µ—И–Є–ї –Љ–Њ–ґ–µ –±–Є вАЮ–љ–∞–є-–≥–Њ–ї—П–Љ–Њ—В–Њ –њ—А–µ–і–Є–Ј–≤–Є–Ї–∞—В–µ–ї—Б—В–≤–Њ –≤ –±–Є–Њ–ї–Њ–≥–Є—П—В–∞вАЬ
–Ґ–µ—Е–љ–Њ–ї–Њ–≥–Є–Є
–Э–µ–≤—А–Њ–љ–љ–∞—В–∞ –Љ—А–µ–ґ–∞ –љ–∞ DeepMind –њ–Њ–±–µ–і–Є –њ—А–Њ—Д–µ—Б–Є–Њ–љ–∞–ї–љ–Є –Є–≥—А–∞—З–Є –љ–∞ StarCraft II (–≤–Є–і–µ–Њ)
















–Ъ–Њ–Љ–µ–љ—В–∞—А–Є
–Ь–Њ–ї—П, —А–µ–≥–Є—Б—В—А–Є—А–∞–є—В–µ —Б–µ –Њ—В T–£–Ъ!
–Р–Ї–Њ –≤–µ—З–µ –Є–Љ–∞—В–µ —А–µ–≥–Є—Б—В—А–∞—Ж–Є—П, –љ–∞—В–Є—Б–љ–µ—В–µ –Ґ–£–Ъ!
–Э—П–Љ–∞ –Ї–Њ–Љ–µ–љ—В–∞—А–Є –Ї—К–Љ —В–∞–Ј–Є –љ–Њ–≤–Є–љ–∞ !
–Я–Њ—Б–ї–µ–і–љ–Є –Ї–Њ–Љ–µ–љ—В–∞—А–Є
4I4ATA
–Э–Њ–≤, —Б–Ї—А–Є—В –і–Њ—Б–µ–≥–∞ –≤—Е–Њ–і –≤ –њ–Є—А–∞–Љ–Є–і–∞—В–∞ –љ–∞ –Ь–Є–Ї–µ—А–Є–љ –µ –Њ—В–Ї—А–Є—В –њ–Њ –∞–љ–Њ–Љ–∞–ї–Є–Є –њ—А–Є —Б–Ї–∞–љ–Є—А–∞–љ–µ
YKoshev
–Я—А–µ—Б—В–Є–ґ–љ–∞ –љ–∞–≥—А–∞–і–∞ –Њ—В –С–Р–Э —Б–њ–µ—З–µ–ї–Є –≥–ї–∞–≤–љ–Є—П—В —А–µ–і–∞–Ї—В–Њ—А –љ–∞ –Э–∞—Г–Ї–∞OFFNews
Johnny B Goode
–Я—А–µ—Б—В–Є–ґ–љ–∞ –љ–∞–≥—А–∞–і–∞ –Њ—В –С–Р–Э —Б–њ–µ—З–µ–ї–Є –≥–ї–∞–≤–љ–Є—П—В —А–µ–і–∞–Ї—В–Њ—А –љ–∞ –Э–∞—Г–Ї–∞OFFNews
Gunteer
–Я—А–µ—Б—В–Є–ґ–љ–∞ –љ–∞–≥—А–∞–і–∞ –Њ—В –С–Р–Э —Б–њ–µ—З–µ–ї–Є –≥–ї–∞–≤–љ–Є—П—В —А–µ–і–∞–Ї—В–Њ—А –љ–∞ –Э–∞—Г–Ї–∞OFFNews