
Докато през юли хората по света се възхищаваха на най-подробните снимки на космоса, направени от космическия телескоп "Джеймс Уеб", биолозите се запознаваха за първи път с друг набор от изображения - такива, които биха могли да направят революция в изследванията на науките за живота.
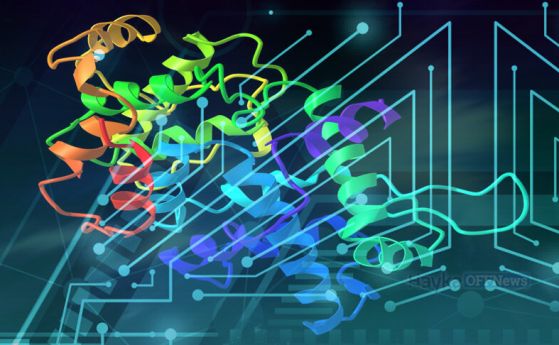
Изображенията представляват предсказаните триизмерни форми на повече от 200 милиона протеина, създадени от система с изкуствен интелект, наречена AlphaFold.
"Можете да си представите, че тя покрива цялата протеинова вселена", заявява Демис Хасабис (Demis Hassabis) на брифинг на 26 юли.
Хасабис е съосновател и главен изпълнителен директор на DeepMind, компанията, създала системата. Комбинирайки няколко техники за дълбоко обучение, компютърната програма е обучена да предсказва формите на протеините, като разпознава модели в структури, които вече са били решени чрез десетилетия експериментална работа с помощта на електронни микроскопи и други методи.
Първата изява на изкуствения интелект става през 2021 г. с прогнози за 350 000 протеинови структури - включително почти всички известни човешки протеини. DeepMind си партнира с Европейския институт по биоинформатика към Европейската лаборатория по молекулярна биология, за да направи структурите достъпни в публична база данни.
Новото обширно издание през юли разшири библиотеката до "почти всеки организъм на планетата, чийто геном е секвениран", посочва Хасабис. "Можете да потърсите триизмерна структура на протеин почти толкова лесно, колкото и при търсене на ключови думи в Google."
Това са прогнози, а не реални структури. Но трябва да се отбележи, че вече изследователите са използвали някои от прогнозите от 2021 г., за да разработят потенциални нови ваксини срещу малария, да подобрят разбирането за болестта на Паркинсон, да разберат как да защитят здравето на медоносните пчели, да получат представа за човешката еволюция и др. DeepMind е насочила AlphaFold и към пренебрегвани тропически болести, включително болестта на Чагас и лайшманиозата, които могат да бъдат инвалидизиращи или смъртоносни, ако не се лекуват.
Публикуването на огромния набор от данни бе посрещнато с вълнение от много учени. Но други се притесняват, че изследователите ще приемат предсказаните структури за истинските форми на протеините. Все още има неща, които AlphaFold не може да прави - и не е бил проектиран да прави - и които трябва да бъдат решени, преди протеиновият космос да придобие напълно ясен облик.
Прогнозите са по-точни за някои протеини, отколкото за други. Грешните прогнози могат да накарат някои учени да си мислят, че разбират как работи даден протеин, докато всъщност не го разбират. Според някои специалисти щателните експерименти продължават да са от решаващо значение за разбирането на начина на сгъване на протеините.
"Сега съществува усещането, че хората не трябва да правят експериментално определяне на структурата, което не е вярно", предупреждава Джули Форман-Кей (Julie Forman-Kay), протеинов биофизик в Детската болница и Университета на Торонто.
Тромави стъпки
Протеините започват като дълги вериги от аминокиселини и се сгъват в множество криволици и други триизмерни форми. Някои от тях приличат на стегнати спираловидни пръстени, на подобни на тирбушон участъци или на гънки на акордеон.
Архитектурата на даден протеин е нещо повече от естетика - тя може да определи как функционира този протеин. Например протеините, наречени ензими, се нуждаят от джоб, в който могат да улавят малки молекули и да извършват химични реакции. А протеините, които работят в протеинов комплекс - два или повече протеина, които си взаимодействат като части на машина - се нуждаят от правилните форми, за да се свържат с партньорите си.
Познаването на гънките, намотките и примките на формата на даден протеин може да помогне на учените да разгадаят как например една мутация променя тази форма, предизвиквайки заболяване. Това знание може да помогне на изследователите да създадат по-добри ваксини и лекарства.
От години учените бомбардират протеинови кристали с рентгенови лъчи, замразяват клетки и ги изследват под мощни електронни микроскопи, както и използват други методи, за да открият тайните на протеиновите форми. Тези експериментални методи отнемат "много време на персонала, много усилия и много пари. Така че това става бавно", разказва Тамир Гонен (Tamir Gonen), мембранен биофизик и изследовател от Медицинския институт "Хауърд Хюз" към Училището по медицина "Дейвид Гефен" в Калифорнийския университет.
Подобна педантична и скъпоструваща експериментална работа е разкрила триизмерните структури на повече от 194 000 протеина, чиито файлове с данни се съхраняват в Protein Data Bank, поддържана от консорциум от изследователски организации. Но ускоряването на темпото, с което генетиците дешифрират ДНК инструкциите за създаване на протеини, далеч надхвърля възможностите на структурните биолози да се справят с него, отбелязва системният биолог Назим Буата (Nazim Bouatta) от Медицинския факултет на Харвард.
"Въпросът на структурните биолози беше: как да преодолеем изоставането?", споделя изследователят.
Мечтата на много изследователи е да разполагат с компютърни програми, които да могат да изследват ДНК на даден ген и да предсказват как кодираният от него протеин ще се сгъне в триизмерна форма.
Тук се намесва AlphaFold
В продължение на много десетилетия учените постигат напредък към тази цел на изкуствения интелект. Но "допреди две години бяхме много далеч от добро решение", разказва Джон Моулт (John Moult), компютърен биолог в кампуса на Университета на Мериленд в Роквил.
Мулт е един от организаторите на състезанието: Critical Assessment of protein Structure Prediction или CASP (Критична оценка на прогнозирането на протеиновата структура). Организаторите дават на участниците набор от протеини, които техните алгоритми трябва да сгънат, и сравняват прогнозите на машините с експериментално определени структури. Повечето изкуствени интелекти не успяват да улучат истинските форми на протеините.
След това през 2020 г. се прояви AlphaFold, който предсказва структурите на 90% от тестовите протеини с висока точност, включително две трети от тях с точност, съперничеща на експерименталните методи.
Разшифроването на структурата на единични протеини е в основата на състезанието CASP от самото му създаване през 1994 г. Благодарение на резултатите на AlphaFold "изведнъж това е направено по същество", добавя Мулт.
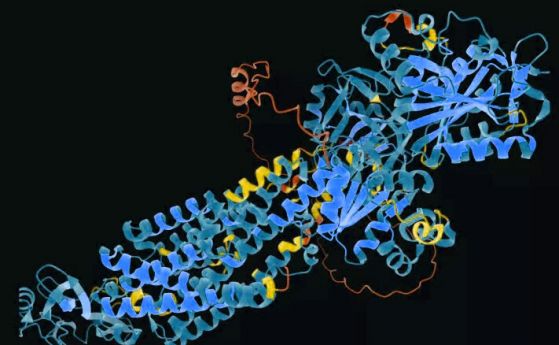
От представянето на AlphaFold през 2021 г. досега повече от половин милион учени са получили достъп до базата данни, отбеляза Хасабис на брифинг. Някои изследователи, например, са използвали прогнозите на AlphaFold, за да им помогнат да се доближат до завършването на огромен биологичен пъзел: ядрения порест комплекс. Ядрените пори са ключови портали, които позволяват на молекулите да влизат и излизат от клетъчните ядра. Без порите клетките не биха работили правилно. Относително всяка пора е огромна, съставена от около 1000 части от около 30 различни белтъка. Преди това изследователите бяха успели да поставят около 30% от парчетата в пъзела.
Този пъзел вече е почти на 60% завършен, след като AlphaFold комбинира предсказанията с експериментални техники, за да разбере как парчетата пасват заедно, съобщават изследователите в Science от 10 юни.
Сега, когато AlphaFold до голяма степен е решил как да се сгъват единични протеини, тази година организаторите на CASP канят екипите да работят по следващите предизвикателства: Предвиждане на структурата на молекулите на РНК и моделиране на взаимодействието на протеините помежду им и с други молекули.
За тези видове задачи, уточнява Мулт, методите на изкуствения интелект с дълбоко обучение "изглеждат обещаващи, но все още не са постигнали резултати".
Къде изкуственият интелект не успява
Възможността да се моделират взаимодействията между протеините би била голямо предимство, тъй като повечето протеини не работят изолирано. Те работят с други протеини или други молекули в клетките. Но точността на AlphaFold при предсказването на това как формите на два протеина могат да се променят при взаимодействието им "не е близо до" точността на неговите точни прогнози за множество единични протеини, подчертава Форман-Кей, протеинов биофизик от Университета в Торонто. Това е нещо, което признават и създателите на AlphaFold.
Изкуственият интелект се обучава да сгъва протеини, като изследва контурите на известни структури. А експериментално са решени много по-малко многопротеинови комплекси, отколкото единични протеини.
Форман-Кей изучава протеини, които не могат да бъдат ограничени в определена форма. Тези вътрешно оплетени протеини обикновено са с формата на мокра юфка. Някои от тях се сгъват в определени форми, когато взаимодействат с други протеини или молекули. И те могат да се сгъват в нови форми, когато се свързват с различни протеини или молекули, за да изпълняват различни задачи.
Предсказаните от AlphaFold форми достигат високо ниво на достоверност за около 60 % от къдравите протеини, които Форман-Кей и колегите му са изследвали, съобщава екипът в предварително проучване, публикувано през февруари в bioRxiv.org. Често програмата изобразява променящите се форми като дълги подобни на тирбушони спирали, наречени алфа спирали.
Групата на Форман-Кей сравнява предсказанията на AlphaFold за три заплетени протеина с експерименталните данни. Структурата, която изкуственият интелект приписва на протеин, наречен алфа-синуклеин, наподобява формата, която протеинът приема, когато взаимодейства с липиди, установява екипът. Но това не е начинът, по който обикновено изглежда протеинът.
За друг протеин, наречен еукариотски протеин 2, свързващ фактор 4Е за иницииране на транслацията, AlphaFold предсказва смесица от двете форми на протеина, когато работи с два различни партньори. Тази Франкенщайнова структура, която не съществува в реалните организми, може да заблуди изследователите за това как работи протеинът, отбелязват Форман-Кей и колегите му.
AlphaFold може би е твърде ограничен в прогнозите си. Една статична "структура не ви казва всичко за това как работи един протеин", обяснява Джейн Дайсън (Jane Dyson), структурен биолог в Изследователския институт "Скрипс" в Ла Джола, Калифорния. Дори отделни протеини с общо взето добре дефинирана структура не са застинали в пространството. Ензимите, например, претърпяват малки промени във формата си, когато управляват химични реакции.
Ако поискате от AlphaFold да предскаже структурата на ензим, той ще покаже фиксирано изображение, което може да прилича много на това, което учените са определили чрез рентгенова кристалография, посочва Дайсън. "Но [тя] няма да ви покаже нито една от тънкостите, които се променят при различните партньори" при взаимодействието с ензима.
"Динамиката е това, което г-н AlphaFold не може да ви даде", подчертава Дайсън.
Революция в процес
Компютърните визуализации дават на биолозите предимство при решаването на проблеми, като например как дадено лекарство може да взаимодейства с даден протеин. Но учените трябва да помнят едно нещо: "Това са модели", а не експериментално разшифровани структури, отбелязва Гонен от Калифорнийския университет.
Той използва прогнозите за протеини на AlphaFold, за да разбере експерименталните данни, но се опасява, че изследователите ще приемат прогнозите на изкуствения интелект като евангелие. Ако това се случи, "рискът е, че ще става все по-трудно и по-трудно да се обоснове защо е необходимо да се определя експериментално структура". Това би могло да доведе до ограничаване на финансирането и други ресурси за видовете експерименти, необходими за проверка на работата на компютъра и за прокарване на нови пътища, смята Гонен.
Буата от Харвардския медицински факултет е по-оптимистично настроен. Той смята, че изследователите вероятно няма нужда да инвестират експериментални ресурси в типовете протеини, които AlphaFold предсказва добре, което би трябвало да помогне на структурните биолози да преценят къде да вложат времето и парите си.
"Има протеини, за които AlphaFold все още се затруднява", съгласен е Буата. Изследователите трябва да изразходват ресурсите си там. "Може би, ако генерираме повече [експериментални] данни за тези трудни протеини, бихме могли да ги използваме за преквалификация на друга система за изкуствен интелект", която би могла да има още по-добри прогнози.
Той и колегите му вече са преработили AlphaFold, за да създадат версия, наречена OpenFold, която изследователите могат да обучават за решаване на други проблеми, като например тези трудни, но важни протеинови комплекси.
Огромните количества ДНК, генерирани в рамките на проекта "Човешки геном", направиха възможни широк спектър от биологични открития и отвориха нови области на изследване. Наличието на структурна информация за 200 милиона протеини може да бъде също толкова революционно, смята Буата.
В бъдеще, благодарение на AlphaFold и неговите роднини с изкуствен интелект, "ние дори няма да знаем какви въпроси бихме могли да зададем". смята изследователят.
Справка:
DeepMind and EMBL-EBI. AlphaFold predicts structure of almost every catalogued protein known to science. Published July 28, 2022.
S. Mosalaganti et al. AI-based structure prediction empowers integrative structural analysis of human nuclear pores. Science. Vol. 376, June 10, 2022, p. 6598. doi:10.1126/science.abm9506.
K.-T. Ko et al. Structure of the malaria vaccine candidate Pfs48/45 and its recognition by transmission blocking antibodies. bioRxiv.org. May 25, 2022. doi:10.1101/2022.05.24.493318.
T.R. Alderson et al. Systematic identification of conditionally folded intrinsically disordered regions by AlphaFold2. bioRxiv.org. February 18, 2022. doi:10.1101/2022.02.18.481080.
J. Jumper et al. Highly accurate protein structure prediction with AlphaFold. Nature. Vol. 596, July 15, 2021, p. 583. doi:10.1038/s41586-021-03819-2.
M. Baek, et al. Accurate prediction of protein structures and interactions using a three-track neural network. Science. Published online July 15, 2021. doi: 10.1126/science.abj8754.
Източник: Has AlphaFold actually solved biology’s protein-folding problem?, Tina Hesman Saey, Science News
Още по темата
Животът
Разработчиците на AlphaFold печелят наградата от 3 млн долара на Breakthrough prize

Животът
Изкуствен интелект изобретява протеини от нулата по поръчка
Животът
Изкуствен интелект откри структурата на почти всеки протеин, известен на науката



















Коментари
Моля, регистрирайте се от TУК!
Ако вече имате регистрация, натиснете ТУК!
Няма коментари към тази новина !
Последни коментари
Прост Човек
Ново обяснение за гигантските експлодиращи кратери в Сибир
dolivo
Климатичният скептицизъм – най-скъпата лъжа на нашето време
helper68
Използването на смартфон в тоалетната е свързано с 46% по-висок риск от хемороиди
YKoshev
Доколко съвместими са минерално-суровинният отрасъл и чистата околна среда?